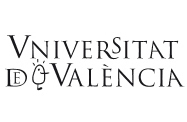
The genome and the complexity of living beings. The genome of an organism is the total dna content of its cells, including genes and intergenic regions. In prokaryotes (Archaea and Bacteria) there is, in general, a linear relationship between genome size and the number of genes. The smallest genomes are found in symbionts and parasites, as they undergo a gene degradation process during adaptation to their new lifestye. However, in eukaryotes there is no correlation between genome size and the complexity of the organism. This is known as the C-value paradox. The largest genome is found in an amoeba, a one-cell organism, with 686.000 Mb, 200 fold larger than the human genome and 20.000 fold larger than the one found in yeast. Now we know that most excess DNA is repetitive DNA, apparently lacking a function (selfish DNA) and whose possible role in genome evolution is still unknown.
Como se sabe, el DNA es el material de los genes; por lo tanto, es natural pensar que organismos más complejos requerirán más genes y tendrán más DNA. Según esto, lo esperado sería: “organismos más complejos tienen genomas de mayor tamaño y contienen un mayor número de genes”. Es decir, a lo largo de la evolución se espera un aumento gradual de los tamaños del genoma y del número de genes.
Para empezar el estudio de los tamaños de genomas, y tratar de comprobar si lo esperado coincide con lo observado, vamos a seguir la clasificación de la vida sobre la tierra, tal y como ha sido propuesta por Woese y colaboradores (1990) a raíz de los estudios filogenéticos realizados con el gen 16S rDNA, que codifica para la subunidad pequeña del RNA ribosómico. Es éste un gen muy conservado en la escala evolutiva y parece que reproduce bien la relación entre los seres vivos. Según la filogenia obtenida, los autores proponen que la vida celular sobre la tierra se puede agrupar en tres dominios: bacterias y arqueas (ambos procariotas) y eucariotas. Una relación simplificada se puede ver en la figura 1.
Rango y tamaños del genoma
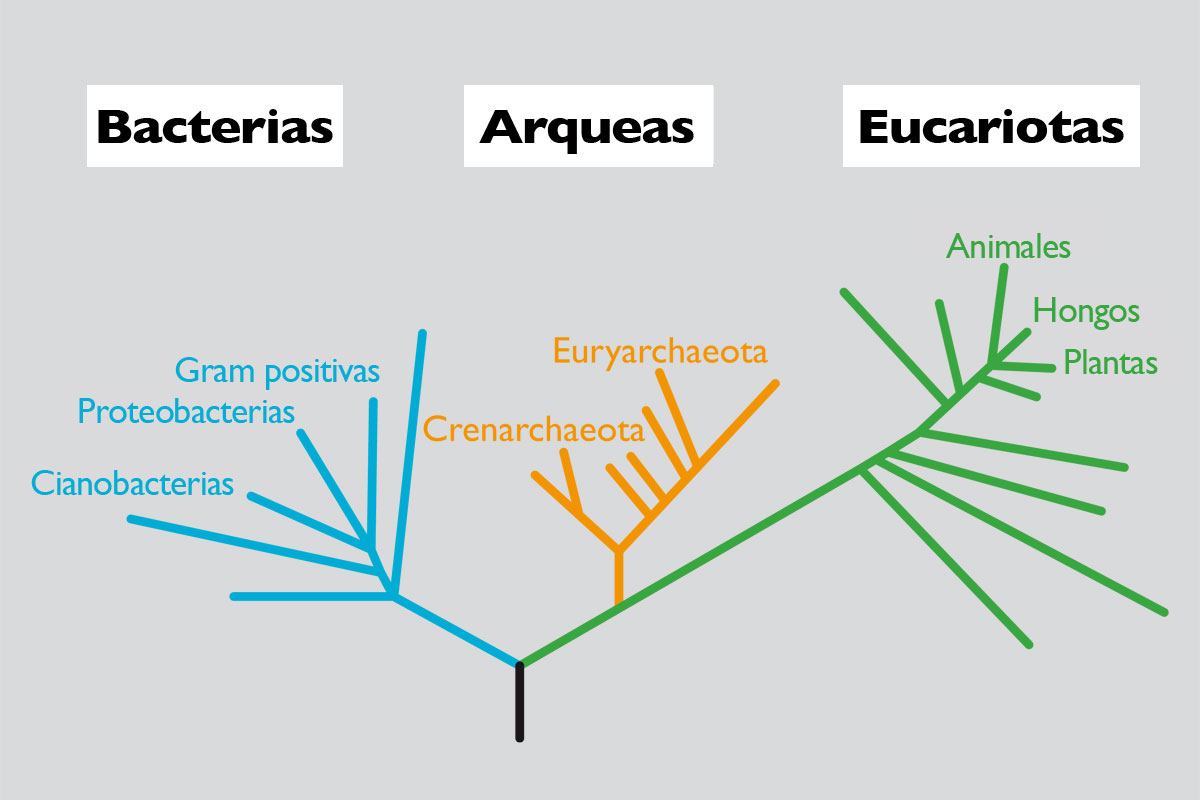
En la figura 2 se representa el rango de tamaños de genomas encontrados en los tres dominios de la vida: bacterias, arqueas y eucariotas. Parece claro que los procariotas tienen, en general, tamaños más pequeños que los eucariotas, con la excepción de algunas bacterias de gran tamaño y algunos eucariotas muy pequeños. Veamos los datos más detalladamente.
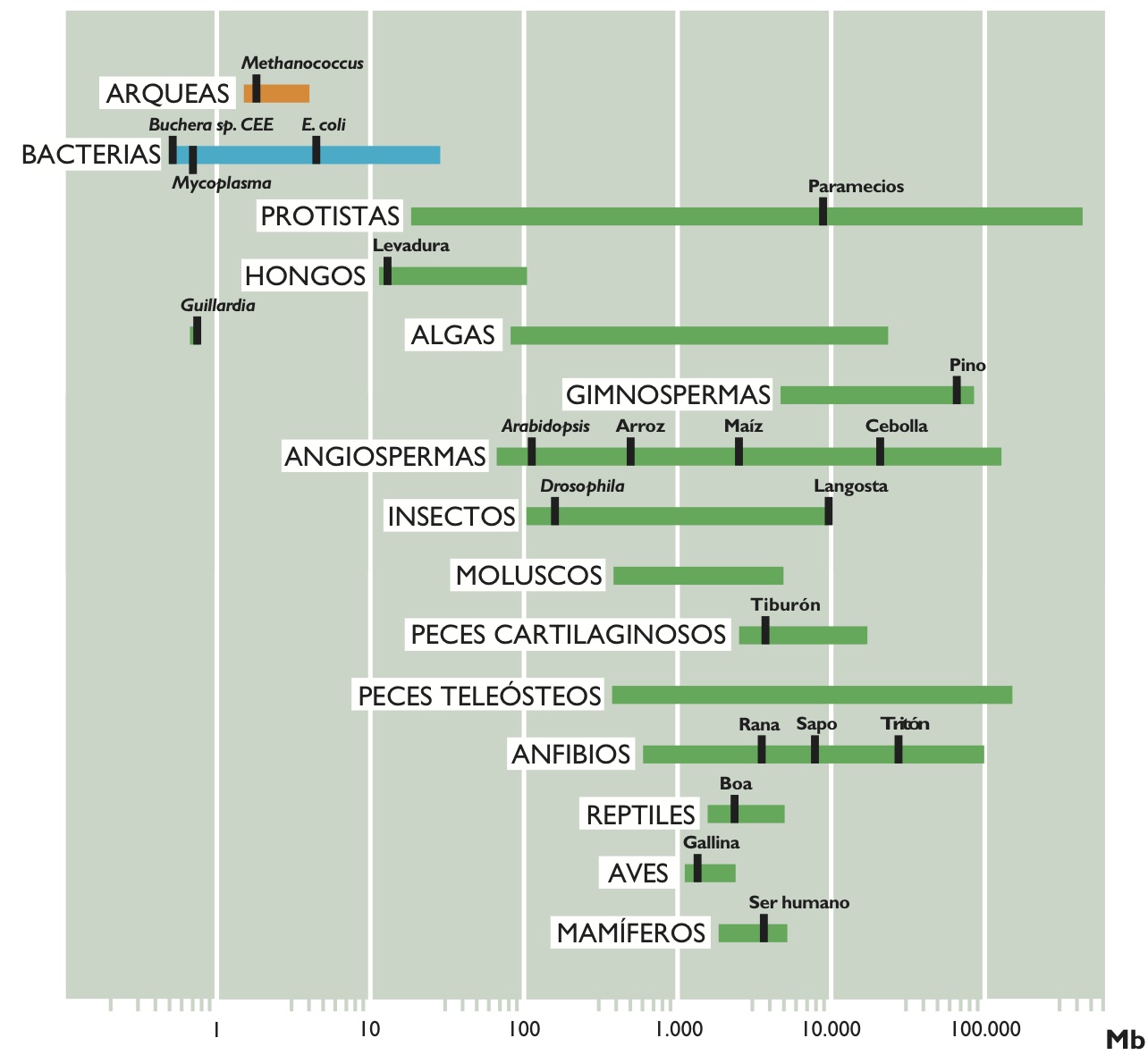
Figura 2. Rango de tamaño del genoma en organismos de los tres dominios de la vida.
Procariotas: bacterias y arqueas
Según los datos publicados hasta el momento, el tamaño varía desde 0,58 megabases (1 megabase (Mb) es un millón de pares de bases (pb)) en el patógeno intracelular Mycoplasma genitalium, hasta más de 10 Mb en varias especies de cianobacterias, con la excepción de Bacillus megaterium, que tiene un genoma de 30 Mb. El segundo genoma más pequeño publicado es el de Buchnera sp. APS, endosimbionte del pulgón de cereales Acyrthosiphon pisum, con un tamaño de 641 kb. Recientemente nuestro grupo de investigación ha caracterizado seis genomas aún más pequeños que el de Mycoplasma, siendo el menor de todos el de Buchnera sp. CCE, endosimbionte del pulgón Cinara cedri, con un tamaño de 0,45 Mb. En general, la mayoría de los genomas tiene un tamaño menor de 5 Mb, como se observa en la tabla 1.
¿Hay una relación entre tamaño de genoma y número de genes? El tamaño del gen procariótico es uniforme, aproximadamente de 900 a 1000 pb. Por tanto, se puede estimar la densidad génica en cada genoma secuenciado. Como se observa en la tabla 1, la densidad génica es más o menos constante, tanto en bacterias, como en arqueas.
Podemos concluir que, al menos en los procariotas, los genomas mayores tienen más genes y son, además, más complejos. Es decir, el número de genes refleja el estilo de vida. Así, bacterias más pequeñas son especialistas, como los parásitos obligados y los endosimbiontes, y bacterias más grandes son generalistas, y pueden incluso tener cierto grado de desarrollo, como es el caso de la esporulación en Bacillus.
Eucariotas: paradoja del valor C
El tamaño del genoma en eucariotas se define como el valor C o cantidad de DNA por genoma haploide, tal como el que existe en el núcleo de un espermatozoide. Se denomina C, por constante o característico, para indicar el hecho de que el tamaño es prácticamente constante dentro de una especie.
Si se observa nuevamente la figura 2, vemos que, en general, los eucariotas tienen genomas mucho mayores que los procariotas, con la excepción de algunas algas verdes endosimbiontes o parásitas, que tienen genomas muy reducidos. En concreto, el genoma eucariótico más pequeño secuenciado es el de Guillardia theta, un alga roja simbionte, de tan sólo 0,55 Mb. También se puede observar en la figura que hay un rango muy amplio de tamaños, mucho mayor que el de procariotas, de hasta más de 80.000 veces, desde organismos como la levadura (1,2 Mb) hasta la ameba (686.000 Mb). Pero ¿existe, como en bacterias, una relación entre el tamaño del genoma y la complejidad del organismo?
En la figura 2 hemos representado el rango de valor C en varios grupos representativos de organismos de eucariotas. Como puede observarse, los protistas unicelulares, como las amebas, muestran la mayor variación en valores C (23,5 Mb a 686.000 Mb, con una razón entre el mayor y el menor de 29.191), mientras que mamíferos, aves y reptiles muestran la menor variación en el tamaño de sus genomas (una razón de sólo 4, 1 y 4, respectivamente). Es más, la gran variación en los tamaños del genoma entre especies eucariotas no parece tener una relación ni con la complejidad del organismo, ni con el número de genes que contienen. Por ejemplo, las amebas, que presentan los mayores genomas, poseen 200 veces más DNA que los humanos (3.400 Mb) y es evidente que una ameba no puede ser más compleja que un humano. Es más, lo esperado sería que los mamíferos, organismos más complejos, presentaran los genomas más grandes. Sin embargo, muchos otros organismos, como peces, anfibios o plantas, tienen genomas mucho mayores que ellos. Incluso cuando comparamos los tamaños entre organismos que parecen similares en cuanto a la complejidad, encontramos también amplias diferencias en sus valores C. Por poner algunos ejemplos, moscas y langostas, cebollas y lirios, etc. tienen variaciones considerables en los tamaños de sus genomas. Los anfibios, como grupo, tienen variaciones de hasta 91 veces y es difícil creer que esto pueda reflejar variaciones de casi 100 veces en el número de genes necesarios para dar lugar a los anfibios correspondientes o que la cebolla necesite 200 veces más DNA que el arroz. La figura 3 representa algunos seres vivos con tamaño proporcional al tamaño de su genoma y no necesita mayor explicación.
Figura 3. Figura 3. Tamaño del genoma en algunos seres vivos. La altura de los dibujos es proporcional al tamaño de su genoma. Los especimenes son la ameba, la cebolla, el saltamontes, el sapo, el ser humano, la gallina, Drosophila y Caenorhabditis, un gusano nematodo.
A la falta de correspondencia entre los valores C y la cantidad presumible de información genética contenida dentro de los genomas se le llamó paradoja del valor C. Puesto que no podemos asumir que una especie posea menos DNA que la cantidad requerida para especificar sus funciones vitales, hemos de explicar por qué muchas especies contienen tal cantidad de exceso de DNA.
DNA génico o DNA no génico
La primera cuestión que se ha de clarificar es si existe una correlación entre el tamaño del genoma y el número de genes. Es decir, ¿las diferencias en los tamaños del genoma son debidas a DNA génico o no génico?
Es sabido, desde finales de los años 60, que el genoma de eucariotas está compuesto de una gran cantidad de DNA repetitivo. Además, desde finales de los años 70 se sabe que los genes se encuentran interrumpidos por secuencias no codificantes, los intrones, que han de ser eliminados antes de que el ribosoma sintetice la proteína. Se trata, en ambos casos de DNA aparentemente superfluo, que contribuye a la gran variación en los valores C y, por tanto, explica la aparente paradoja.
El tamaño y el número de intrones varían ampliamente a lo largo de la escala evolutiva, siendo los mamíferos los que tenemos el mayor número y de mayor tamaño. También el DNA repetitivo varía entre organismos. Tradicionalmente se clasifica este DNA como: altamente repetitivo, con secuencias como los microsatélites y los minisatélites; y moderadamente repetitivo, donde se encuadran los elementos transponibles, las secuencias que constituyen el ejemplo más claro de DNA egoísta.
Número de genes y complejidad del organismo
A medida que se van completando las secuencias de genomas enteros, se va conociendo con mayor o menor exactitud el número de genes que se derivan de esas secuencias, ya que lo que se tenía hasta entonces eran estimaciones indirectas. Sin embargo, algunos datos están deparando sorpresas porque, en algunos casos, no parece haber una correlación clara entre número de genes y complejidad del organismo. El gusano nematodo C. elegans, tiene 18.000 genes (tabla 1), unos 5.000 más que Drosophila, un organismo más complejo. El hombre sólo tiene el doble de genes que C. elegans (las estimaciones indicaban unos 100.000). También estos datos se empiezan a entender. Existen mecanismos en eucariotas superiores que son capaces de “expandir el proteoma”. Es decir, a partir de una misma secuencia de DNA obtener más de una proteína. Los grandes intrones encontrados en mamíferos pueden, en muchos casos, “esconder” información que no se puede deducir sólo con la secuencia del DNA. Aún tiene que pasar tiempo hasta que se pueda conocer el número de proteínas que un organismo es capaz de sintetizar. Pero esto sería motivo de otro trabajo. En cualquier caso, sí que se puede afirmar que “organismos más complejos tienen más funciones génicas”.
| Organismo | ||||
| Nombre común o clase | Nombre científico | Tamaño del genoma (Mb) |
Número de genes |
Densidad génica (genes/Mb) |
| Eucariotes | Saccharomyces cerevisiae | 12 | 6.241 | 480 |
| Levadura del pan | ||||
| Nematodo | Caenorhabditis elegans | 97 | 18.424 | 190 |
| Crucífera | Arabidopsis thaliana | 125 | 25.498 | 204 |
| Mosca del vinagre | Drosophila melanogaster | 180 | 13.601 | 75 |
| Pez globo | Fugu rubripes | 400 | 35.000 | 100 |
| Arroz | Oryza sativa | 450 | ||
| Erizo de mar | Strongylocentrotus purpuratus | 900 | 27.350 | 30 |
| Maíz | Zea mays | 2.400 | ||
| Ser humano | Homo sapiens | 3.400 | 35.000 | 10 |
| Cebolla | Allium cep | 18.000 | ||
| Ameba | Amoeba dubia | 686.000 | ||
| Arqueas | Aeropyrum pernix | 1,55 | 1.522 | 981 |
| Crenarchaeota | ||||
| Euryarchaeota | Methanococcus jannaschii | 1,66 | 1.715 | 1033 |
| Euryarchaeota | Archaeoglobus | 2,18 | 2.420 | 1110 |
| Bacterias | Buchnera sp. CCE | 0,45 | ||
| Proteobacteria | ||||
| Gram positiva | Mycoplama genitalium | 0,58 | 479 | 831 |
| Proteobacteria | Buchnera sp. APS | 0,64 | 564 | 881 |
| Gram negativa | Haemophilus influenzae | 1,8 | 1.727 | 959 |
| Cianobacteria | Synechocystis sp. | 3,6 | 3.168 | 880 |
| Gram positiva | Bacillus subtilis | 4,2 | 4.100 | 976 |
| Proteobacteria | Escherichia coli | 4,6 | 4.288 | 932 |
Tabla 1. Tamaño del genoma, número de genes y densidad génica.
Mecanismos moleculares que alteran el tamaño del genoma
Son muchos los mecanismos mutacionales que pueden producir cambios en el tamaño del genoma. Algunos de ellos ocurren a gran escala (duplicación de todo el genoma), mientras que otros ocurren a una escala muy pequeña (pérdida o ganancia de unos pocos nucleótidos). Sin embargo, hay que destacar que estas mutaciones afectan, en principio, a una sola célula, que en el caso de ser un gameto, podrá transmitir esta mutación a un descendiente. Este individuo tendrá que convivir con otros en una población y sólo en futuras generaciones sabremos si la mutación que lleva se extenderá a todos los individuos de la población (fijación) o, por el contrario, desaparecerá. La probabilidad de que ocurra una u otra cosa dependerá de mecanismos evolutivos como la selección natural (si aporta una ventaja o desventaja al individuo) o la deriva genética (por azar).
Los mecanismos cromosómicos suelen producir variaciones drásticas con una única mutación. Se puede destacar la duplicación de todo el genoma, duplicaciones que afectan a un solo cromosoma, o a una parte de él. De forma equivalente, también se conocen mutaciones que producen la pérdida de algún fragmento cromosómico. Este tipo de cambios, aunque igualmente frecuente en plantas y animales, en las primeras parecen haber contribuido de forma relevante a la evolución de su genoma.
«Se puede afirmar que «organismos más complejos tienen funciones génicas»»
Los elementos genéticos móviles, o elementos transponibles, son otros de los causantes de grandes variaciones en el tamaño del genoma. Estos elementos, de unos pocos miles de nucleótidos, se duplican y las copias duplicadas se insertan en otras partes del genoma, lo que provoca rápidos aumentos en su tamaño, salvo que actúen los mecanismos de control de su proliferación (como auténticos egoístas desarrollan mecanismos para regular su movimiento por el genoma, de manera que no produzcan daños irreparables, ya que ello implicaría su propia desaparición). Se ha estimado que el genoma del maíz se ha duplicado a causa de los elementos transponibles en los últimos tres millones de años de su evolución.
Se considera que las inserciones o deleciones espontáneas (llamadas indels) de unos pocos nucleótidos son una de las causas más importantes de la evolución del tamaño del genoma a largo plazo. En varias especies de insectos, por ejemplo, se ha mostrado una gran correlación entre la tasa global de pérdida de DNA de las regiones intergénicas y no codificantes y el tamaño del genoma. La fijación de estas mutaciones es muy improbable si el indel afecta a un gen, pero es más probable si afecta a pseudogenes (genes no funcionales, inactivados recientemente) o a otras secuencias de DNA sin función. La desintegración de los genes, o desaparición de un gen del genoma, suele transcurrir en un primer paso con su inactivación mediante una mutación puntual (formación de un pseudogén). Posteriormente, el DNA que forma los pseudogenes va siendo eliminado hasta perderse cualquier vestigio de su presencia en el genoma.
La variación en la longitud del DNA de los minisatélites y microsatélites es otro de los mecanismos que pueden alterar el tamaño del genoma. Estas secuencias están formadas por una unidad de pocos nucleótidos, repetida de forma contigua desde menos de diez hasta varios miles de veces. El número de unidades repetidas varía mucho, incluso en individuos de la misma especie. Ello es debido a dos mecanismos que provocan tanto el aumento como la disminución del número de repeticiones. Estos mecanismos son la recombinación, llamada desigual, y errores durante la replicación del DNA, debido al fenómeno conocido como el deslizamiento de la DNA polimerasa. De hecho, éstas son las secuencias que se están utilizando para, por ejemplo, identificar restos humanos, debido a su gran variabilidad.
Uno de los procesos de mayor importancia en el aumento del tamaño del genoma en organismos unicelulares, especialmente procariotas, es la transferencia genética horizontal. Este proceso consiste en la introducción en el genoma de una especie de un fragmento de DNA de otra especie, que contiene uno o varios genes. El DNA se introduce en la célula por diferentes mecanismos y luego ha de recombinar con el genoma. Si los genes introducidos confieren alguna ventaja al organismo suelen mantenerse, pero si esto no ocurre pueden mutar, inactivándose. Un ejemplo de la importancia de estos procesos se puede ver en la bacteria intestinal Escherichia coli, algunas de cuyas cepas se han vuelto patógenas por la transferencia de genes de virulencia. La importancia de este fenómeno en periodos recientes de la evolución de organismos superiores es mucho más limitada, aunque hay muchos casos descritos de transferencia genética intracelular de DNA del genoma de las mitocondrias y cloroplastos al genoma nuclear.
Todas estas mutaciones surgen de forma periódica en multitud de especies, incluida la especie humana. Muchas de ellas son desventajosas y la presión selectiva las va eliminando de la población, otras individualmente son neutras pero pueden ser colectivamente ventajosas o perjudiciales, al aumentar o disminuir el tamaño del genoma. En bacterias, tener un genoma pequeño puede ser positivo para optimizar el tiempo y coste de la duplicación del DNA. En organismos eucariotas, se han propuesto diversas ventajas por tener un tamaño grande del genoma, aunque no vaya asociado a un mayor número de genes.