Si alguna cosa ha quedat clara en l’actual pandèmia protagonitzada per la malaltia COVID-19, és que tot ha ocorregut a una velocitat vertiginosa, també en la ciència. Des que el passat 31 de desembre del 2019 les autoritats de la República Xinesa van alertar l’Organització Mundial de la Salut que estaven tractant desenes de casos de pneumònia d’origen desconegut, científics de tot el planeta es van posar a la feina per a primer, descobrir el patogen que s’ocultava darrere d’aquestes pneumònies, i després, estudiar-lo en detall per a poder desenvolupar alguna estratègia terapèutica que mitigara els seus devastadors efectes.
Així, en les primeres setmanes de gener un grup d’investigadors de la Universitat de Fudan (la Xina) va seqüenciar el genoma del primer SARS-CoV-2, el coronavirus responsable de la nova malaltia, a partir de mostres del fluid de rentat broncoalveolar d’un pacient de 41 anys sense historial clínic d’hepatitis, tuberculosi o diabetis, i ho va alliberar fins i tot abans que fora acceptat per a la seua publicació el 28 de gener (Nature 580, E7 [2020].). A partir d’aquest moment, investigadors de tot el món van iniciar una carrera trepidant per a conèixer els detalls moleculars d’aquest patogen. En l’actualitat, només uns pocs mesos després i amb diversos milers de genomes seqüenciats, s’ha generat una quantitat ingent d’informació certament difícil de manejar.
Organització i expressió del genoma
El genoma del SARS-CoV-2 està format per un RNA de cadena senzilla de vora 29.900 nucleòtids que pot ser reconegut i traduït per la maquinària cel·lular de síntesi de proteïnes de les cèl·lules infectades. Aquest genoma pot codificar fins a 29 proteïnes, si bé no és clar encara si totes aquestes proteïnes se sintetitzen realment durant el cicle vital del virus. De les proteïnes codificades en el genoma quatre d’elles donen forma a l’estructura del coronavirus i per això es denominen proteïnes estructurals. Aquestes quatre proteïnes estructurals són la glicoproteïna de la superfície (Spike, S) que forma les espícules característiques d’aquesta família de virus; la proteïna que forma la matriu de la membrana (Membrane, M) que és la més abundant en l’embolcall lipídic del virus; una xicoteta proteïna també embeguda en la membrana lipídica que es troba en un nombre reduït de còpies (Envelope, E), i la proteïna que s’associa a l’RNA viral estabilitzant-lo i que forma la nucleocàpsida del virus (Nucleocapsid, N).
«Si alguna cosa ha quedat clara en l’actual pandèmia protagonitzada per la malaltia COVID-19, és que tot ha ocorregut a una velocitat vertiginosa, també en la ciència»
Aquestes quatre proteïnes estructurals estan codificades en l’extrem 3’ del genoma (Figura 1) i, igual que altres nou proteïnes accessòries codificades en aquesta regió, no són traduïdes directament pels ribosomes de la cèl·lula infectada, sinó que se sintetitzen a partir d’RNA subgenòmics (sgRNA) generats per una proteïna viral encarregada de replicar (copiar) el genoma del virus (nsp12, veure a baix). En l’actualitat encara no coneixem si totes aquestes proteïnes accessòries se sintetitzen en els pacients CoVid19, i de fet els estudis més recents no han aconseguit detectar tots els sgRNA (Kim et al., The Architecture of SARS-CoV-2 Transcriptome, Cell [2020].) i tampoc hi ha evidències de la presència d’algunes d’aquestes proteïnes en les cèl·lules infectades.
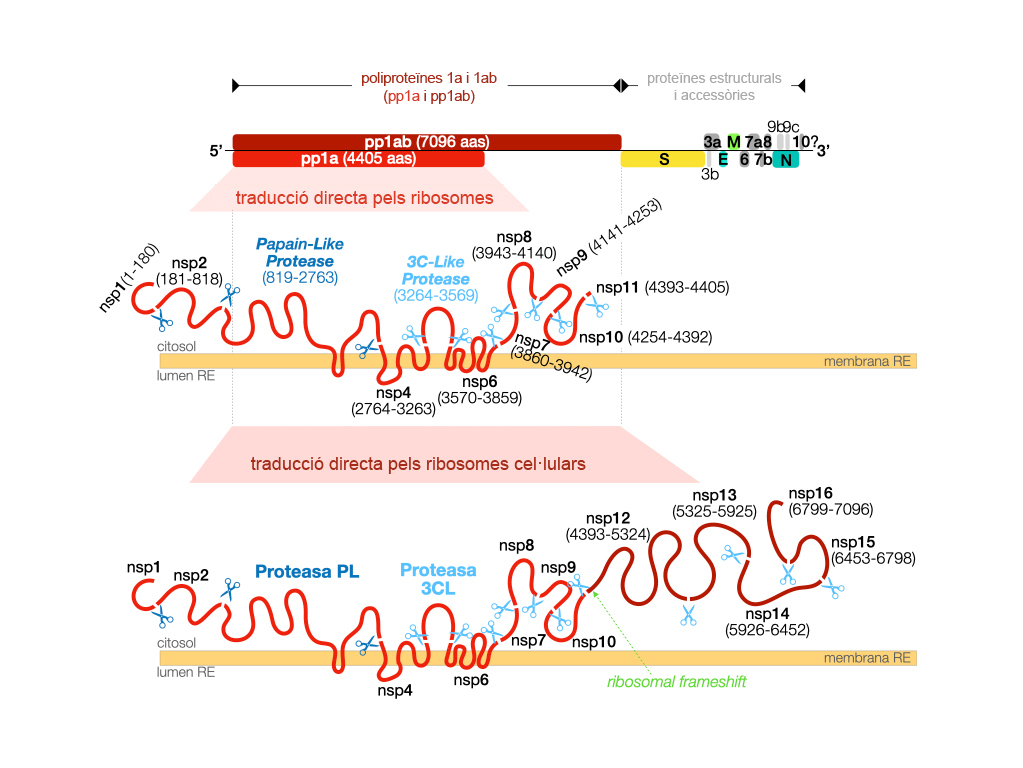
Figura 1. Representació esquemàtica de l’organització del genoma del SARS-CoV-2 i de les proteïnes que codifica, il·lustrant-se amb major detall les poliproteïnes pp1a i pp1ab que contenen les proteases PL i 3CL.
A diferència del que ocorre amb les proteïnes estructurals i accessòries, que necessiten els sgRNAs com a ‘missatgers’ per a la seua síntesi, la resta del genoma del coronavirus codifica en el seu extrem 5’ (fet que no és casual) dues poliproteïnes que sí que poden ser sintetitzades directament a partir de l’RNA genòmic pels ribosomes de la cèl·lula infectada. Això vol dir que aquesta regió, que representa aproximadament el 67% del genoma del virus, s’organitza com dues pautes de lectura obertes (ORF1a i ORF1ab, vegeu la Figura 1) que codifiquen diverses proteïnes que no estan separades entre elles, sinó que se sintetitzen com a llargues cadenes d’aminoàcids que hauran de ser digerides (tallades) per a generar 16 proteïnes no estructurals (non-structural proteins, nsp). Com veurem, aquest fet diferencial en el cicle del virus constitueix un dels seus possibles talons d’Aquil·les.
Les regions codificants de les ORF1a i ORF1ab comparteixen la mateixa informació en el seu extrem 5’, de manera que quan els ribosomes s’uneixen al RNA viral sintetitzen la poliproteïna de membrana 1a (polyprotein1a, pp1a) en l’entorn del reticle endoplàsmic de la cèl·lula infectada. Com hem assenyalat, aquesta gran poliproteïna de 4405 aminoàcids ha de tallar-se per a donar lloc a les proteïnes funcionals del virus. Per a això han de trencar-se els enllaços químics, enllaços peptídics, que conformen l’esquelet de les proteïnes. En condicions normals aquest procés és extraordinàriament lent, la qual cosa explica que les proteïnes siguen estables en els éssers vius (el temps necessari per al trencament d’enllaços peptídics a temperatura ambient és de diversos centenars d’anys).
«Per a poder desenvolupar fàrmacs antivirals eficaços és necessari conèixer en detall l’estructura i el mecanisme d’acció de les proteases»
Per a dur a terme aquest trencament a una velocitat compatible amb el cicle de replicació del virus, és necessària l’acció d’enzims, unes proteïnes especialitzades a accelerar reaccions químiques. El virus codifica dues d’aquestes proteïnes que reben el seu nom per la funció que realitzen: proteases, trencament de proteïnes. La principal d’aquestes proteases, la 3CL (3Chymotrypsin-Like), forma part de la pp1a i una vegada sintetitzada s’autoproteolitza (es talla a ella mateixa) i en la seua forma madura realitza 5 talls addicionals en la seqüència de la poliproteïna per a generar les proteïnes no estructurals nsp4-nsp11. A més d’aquesta proteasa, els coronavirus en codifiquen una altra denominada PL (Papain-Like), que dificulta la resposta cel·lular enfront del virus i realitza 3 talls a la pp1a generant les nsp1, nsp2 i a ella mateixa.
El final de la seqüència que codifica la pp1a presenta un senyal (un codó) de parada que indica al ribosoma la fi de la síntesi de la poliproteïna. No obstant això, en aproximadament el 60% dels casos, els ribosomes en traduir aquesta regió del RNA viral poden patir un canvi en la pauta de lectura (ribosomal frameshift), desapareixent d’aquesta manera el senyal de parada. Aquest canvi permet als ribosomes llegir més enllà de la regió que codifica la pp1a, la qual cosa condueix a la síntesi de la pp1ab. La pp1ab és una poliproteïna enorme (7096 aminoàcids) que inclou a més de les proteïnes no estructurals descrites en la pp1a entre altres l’enzim que copia el genoma del virus (RNA polimerasa RNA dependent, nsp12), del qual hem parlat anteriorment.
Inhibint les proteases
Com podem imaginar, l’activitat d’ambdues proteases resulta crucial per a la replicació del virus, per la qual cosa han emergit com unes de les dianes preferents per al desenvolupament de fàrmacs que ens permeten tractar aquesta terrible malaltia. Per a poder desenvolupar fàrmacs antivirals eficaços és necessari conèixer en detall l’estructura i el mecanisme d’acció de les proteases. De nou en temps rècord (al gener!) vam disposar de les primeres estructures d’alta resolució de la proteasa 3CL (Science 24 Apr 2020:Vol. 368, Issue 6489, pp. 409-412; y Nature [2020].). La rapidesa d’aquest notable èxit per part de dos grups de recerca de manera independent (un a Berlín i un altre a Xangai) es va deure en gran part a l’experiència que tots dos grups van adquirir treballant amb el coronavirus SARS-CoV, causant de l’epidèmia de la síndrome respiratòria aguda greu que va aparèixer en el sud-est asiàtic entre els anys 2002 i 2004. Les proteïnes del SARS-CoV-2 presenten una gran similitud amb les d’aquest altre coronavirus (fins al 96% en el cas de la proteasa principal) i amb les del coronavirus causant del brot de la síndrome respiratòria de l’Orient Mitjà (MERS).
Resulta si més no curiós que línies d’investigació que podrien haver-se considerat de poca actualitat fa només uns mesos a causa de la desaparició dels dos brots esmentats, hagen resultat ara claus per a poder abordar d’una forma ràpida l’estudi del SARS-CoV-2. Aquest fet ens adverteix, una vegada més, del perill que la ciència s’oriente únicament per criteris d’aparent utilitat o aplicabilitat immediata.
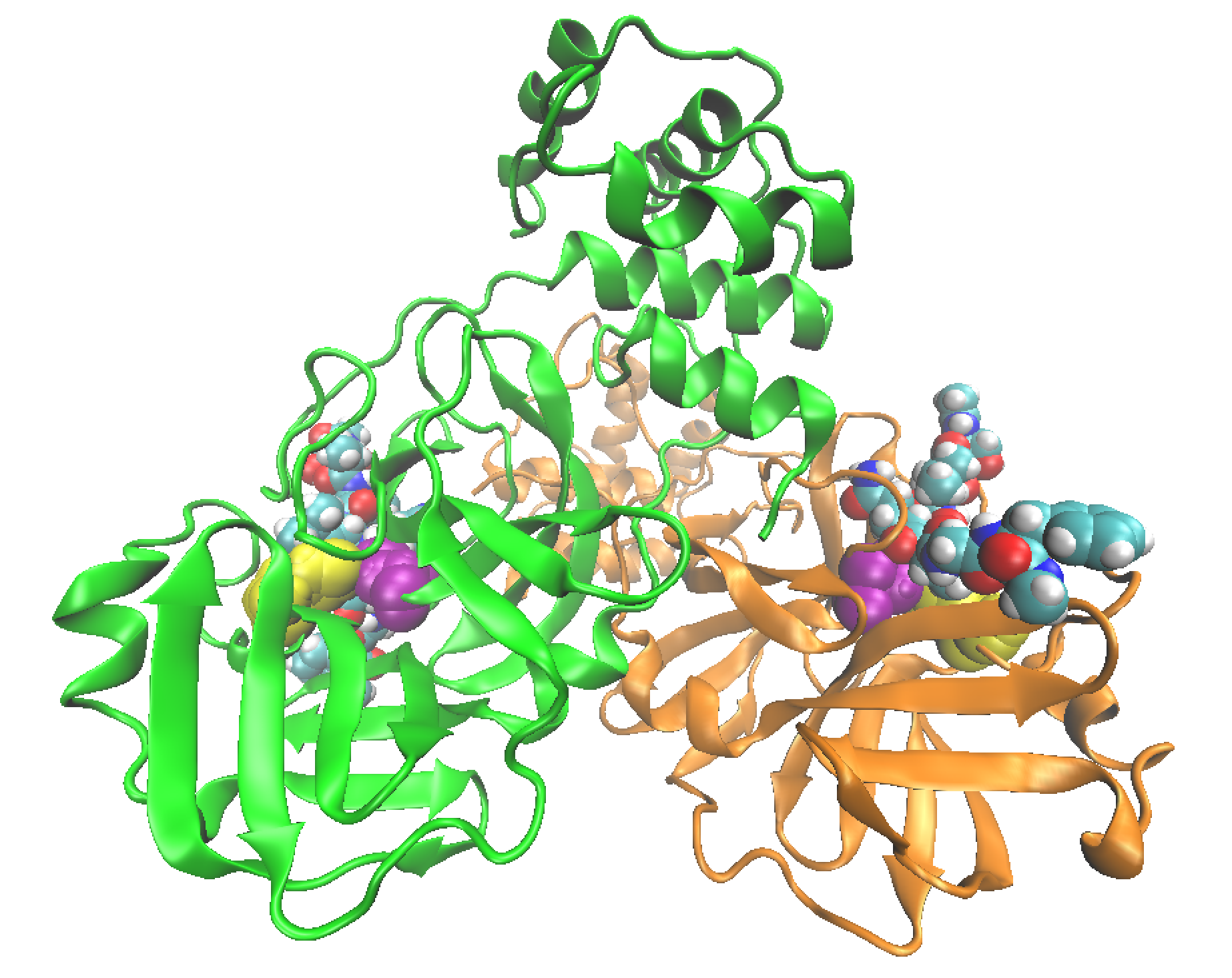
Figura 2. Representació de l’estructura de la proteasa 3CL-Like. Cadascun dels protòmers apareix representat en un color amb un pèptid substrat (representat en forma d’esferes per a cadascun dels seus àtoms) en cadascun dels centres actius. La cisteïna i histidina catalítiques apareixen representades com a esferes en color groc i porpra respectivament en cadascun dels protòmers.
La 3C-Like proteasa del virus és un dímer, format per dues subunitats idèntiques, tal com pot observar-se en la Figura 2. La formació del dímer és essencial per a la seua activitat, ja que el centre actiu, el lloc concret on la proteasa s’uneix amb la poliproteïna substrat i duu a terme la seua activitat, queda conformat completament quan es produeix la interacció entre les dues subunitats. Cadascun dels dos centres actius del dímer conté una parella d’aminoàcids encarregats de dur a terme el trencament d’enllaços peptídics en les poliproteïnes, la diada catalítica formada per una cisteïna, Cys145, i una histidina, His41 (vegeu el vídeo adjunt).
La proteasa principal del virus presenta una gran especificitat per a trencar l’enllaç peptídic format entre un residu de glutamina i un altre aminoàcid de xicoteta mida (glicina, alanina o serina). Cap de les proteases conegudes en humans presenta aquesta especificitat, la qual cosa converteix la proteasa principal del SARS-CoV-2 en una diana terapèutica excel·lent, ja que un fàrmac dissenyat per a inhibir la seua activitat tindrà baixes probabilitats d’interaccionar amb proteases humanes i, per tant, de presentar efectes secundaris. Aquests inhibidors són molècules capaces d’unir-se al centre actiu de la proteasa impedint l’accés de les poliproteïnes i que, per tant, la proteasa realitze la seua funció, la qual cosa implica la interrupció del cicle de replicació del virus. De fet, la inhibició de proteases principals de virus és ja una estratègia eficient que s’usa en el tractament d’altres epidèmies, com és el cas de la SIDA.
Com hem assenyalat abans, el disseny d’inhibidors per a la 3C-Like proteasa del SARS-CoV-2 s’està duent a terme partint del coneixement adquirit sobre la inhibició del seu enzim equivalent en el SARS-CoV. El grup de Berlín (Science 24 Apr 2020:Vol. 368, Issue 6489, pp. 409-412) va utilitzar la informació estructural obtinguda per a la proteasa principal del SARS-CoV-2 per a optimitzar un inhibidor que ja s’havia mostrat efectiu contra les proteases principals del SARS-CoV i del MERS-CoV. Aquest inhibidor és capaç d’unir-se de manera irreversible al centre actiu de la proteasa, formant un enllaç estable amb la cisteïna catalítica, deixant-la d’aquesta manera inservible per a realitzar la seua funció sobre les poliproteïnes.
Per part seua, el grup de Xangai va obtenir l’estructura de la proteasa principal juntament amb un inhibidor diferent unit també a la cisteïna catalítica (Nature. [2020].). Aquest inhibidor s’havia desenvolupat també contra les proteases principals del SARS-CoV i del MERS-CoV. A més, l’equip de Xangai va rastrejar entre fàrmacs coneguts i productes naturals amb l’objectiu de seleccionar altres possibles punts de partida per a desenvolupar inhibidors més eficaços. Així, van seleccionar trenta candidats; les seues estructures en complex amb la proteasa s’estan resolent en el Diamond Light Source, un laboratori sincrotró situat al Regne Unit. Aquest laboratori ha posat en marxa una interessant iniciativa fent accessibles al públic totes les estructures de complexos proteasa-inhibidor resoltes i obrint una pàgina web en la qual és possible suggerir possibles inhibidors que, després de ser avaluats mitjançant intel·ligència artificial, són assajats després de prioritzar-se sobre la base de criteris com ara la seua facilitat de síntesi o la seua possible toxicitat.
«L’obtenció d’un fàrmac capaç d’inhibir alguna de les proteases del SARS-CoV-2, o de qualsevol altre agent patogen, és un procés llarg i complex que exigeix la participació de diferents branques de la ciència»
La proteasa PL (Papain-Like) conté també en el seu centre actiu una cisteïna i una histidina encarregades de dur a terme de trencament de l’enllaç peptídic. Per tant, el mecanisme emprat per a actuar com proteasa és molt similar a l’empleat per la 3C-Like. No obstant això, la proteasa PL actua específicament sobre enllaços peptídics situats després de dues glicines consecutives, usant una seqüència de reconeixement que, desafortunadament, també és emprada per algunes proteases humanes. Aquesta coincidència de seqüència de reconeixement fa més difícil el disseny d’inhibidors específics per a la proteasa PL del virus que no causen, al seu torn, efectes secundaris en interferir també l’activitat de proteases humanes. De nou en aquest cas els estudis realitzats sobre la proteasa PL del SARS-CoV o del MERS-CoV, molt similars a la del SARS-CoV-2, poden facilitar el desenvolupament d’inhibidors específics per a aquesta proteasa.
Malgrat tots els assoliments aconseguits en tan curt termini de temps, és necessari subratllar que l’obtenció d’un fàrmac capaç d’inhibir alguna de les proteases del SARS-CoV-2, o de qualsevol altre agent patogen, és un procés llarg i complex que exigeix la participació de diferents branques de la ciència. El procés pot començar per la determinació de l’estructura de la proteïna diana, seguit del disseny assistit per ordinador de molècules candidates a inhibir la seua activitat, la síntesi química, la realització de proves bioquímiques sobre la seua activitat in vitro i, finalment, l’estudi de la viabilitat com a fàrmac administrat primer a cèl·lules, després a animals d’experimentació i finalment a humans. Aquest últim pas és fonamental per a comprovar els possibles efectes secundaris que l’inhibidor pot causar sobre el nostre organisme.
Els terminis poden acurtar-se molt significativament si l’inhibidor és un fàrmac ja aprovat per al tractament d’altres malalties. Aquesta és la raó que explica el perquè s’estan realitzant proves massives in vitro i in vivo de diferents fàrmacs per a inhibir alguna de les proteïnes del SARS-CoV-2, inclosa la proteasa. Així, el grup de Xangai ha publicat recentment una estructura de la proteasa principal del SARS-CoV-2 inhibida per carmofur, un antineoplàstic utilitzat per a combatre càncers colorectals i que podria ser un punt de partida prometedor per al desenvolupament de nous antivirals. Quan podrem disposar d’ells només la ciència (i el seu finançament) ho dirà.
Representació de l’atac nucleofílic del Sγ (en groc) de la cisteïna-145 de la proteasa 3CL sobre l’enllaç peptídic de la poliproteïna substrat (la direcció de l’atac s’ha destacat com a línia de punts). Prèviament, la histidina-41 ha transferit un àtom d’hidrogen (representat en blanc i amb una línia de punts) a l’àtom de nitrogen de l’enllaç peptídic que es trencarà. El vídeo és cortesia de Carlos A. Ramos-Guzmán, investigador de la Universitat de València.