Rere les petjades del nostre passat
Paleoproteòmica, l’inici d’una nova era?

Per les restes trobades, es pensa que Gigantopithecus blacki podia arribar als tres metres d’alçada i pesar al voltant de 300 kg. Però la manca de fòssils impedia a la comunitat científica saber-ne més, d’aquest primat gegant extint. Ara, gràcies a la paleoproteòmica, s’ha pogut comparar la seqüència de proteïnes d’aquesta espècie amb la d’altres grans simis i establir que Gigantopithecus era un parent proper dels orangutans. Dalt, representació artística de Gigantopithecus blacki. / Ikumi Kayama (Studio Kayama LLC)
El 1935 l’antropòleg alemany Gustav von Koenigswald va adquirir unes misterioses dents en apotecaries del sud de la Xina. Les dents, d’uns 2,5 centímetres, s’utilitzaven en la medicina tradicional xinesa i eren anomenades «dents de drac». Von Koenigswald va quedar perplex per la seva mida desmesurada. Les dents mostraven trets característics dels primats, però eren més grans que els molars dels goril·les, els primats actuals més grans del planeta. No és sorprenent, doncs, que Von Koenigswald anomenés la nova espècie Gigantopithecus. A mitjan segle XX, noves troballes de fòssils van consolidar aquesta espècie com un llinatge de primats que va habitar el sud-est asiàtic des de fa uns dos milions d’anys fins fa 100.000 anys, amb la incertesa de fins a quin punt formava part dels nostres orígens. En l’actualitat s’han trobat quatre mandíbules i prop de 1.300 dents. Malgrat que la mida del gegant només s’ha pogut extrapolar a partir d’ossos del crani, se suposa que Gigantopithecus podia arribar als tres metres d’alçada i pesar al voltant de 300 kilograms (Zhang i Harrison, 2017), el primat més gran mai descobert.
La comunitat científica no va poder aprendre gaire més sobre Gigantopithecus a causa de la manca de fòssils com ara cranis complets i esquelets. Sense dades moleculars, com ara la seqüència de proteïnes o d’ADN, les relacions de fòssils i altres organismes només es poden investigar mitjançant comparacions morfològiques. De fet, fins fa poques dècades el nostre coneixement sobre la història dels homínids es basava en la comparació d’estructures òssies entre fòssils. Malgrat que els estudis paleontològics han estat i són cabdals per a entendre el passat de la nostra espècie, tenen certes limitacions a l’hora de resoldre posicions filogenètiques o caracteritzar en detall esdeveniments demogràfics.
De la paleogenòmica a la paleoproteòmica
Actualment, gràcies a grans avenços tecnològics, podem obtenir dades moleculars d’organismes que van viure fa desenes de milers d’anys mitjançant la seqüenciació del seu ADN. Aquesta «nova habilitat», la d’obtenir dades moleculars d’organismes fossilitzats, ha obert un gran ventall de possibilitats i ha permès explorar preguntes abans impossibles de respondre. Per exemple, gràcies a l’estudi de l’ADN de neandertals i denissovans, els nostres cosins més propers, sabem que el seu llinatge va divergir del nostre fa uns 500.000 anys (Prüfer et al., 2014). També sabem que totes les poblacions humanes fora de l’Àfrica tenen prop d’un 3 % d’ADN neandertal i que algunes poblacions d’Oceania mostren, a més de l’ADN de neandertal, fins a un 5 % de material genètic provinent de denissovans (Reich et al., 2010), molt probablement com a resultat de l’intercanvi de gens entre els nostres ancestres.

Entrada de la cova de Chuifeng, a la Xina, on es van trobar les restes de Gigantopithecus de les quals ha estat possible obtenir proteïnes per a ser analitzades. La seua antiguitat, d’uns 1,9 milions d’anys, no permetia extraure’n l’ADN. / Wei Wang
Així doncs, si podem obtenir l’ADN d’organismes fossilitzats, com és que la comunitat científica no seqüencia tot el que troba? La resposta és força intuitiva. Imagineu la Terra fa uns 1,9 milions d’anys, temps en el qual les restes d’un o una Gigantopithecus blacki van acabar en una cova al sud de la Xina, la cova de Chuifeng. Potser l’animal va morir allà, o bé algun carronyaire va transportar part de les seves restes fins a la cova. En poc temps, la carn va desaparèixer i l’esquelet va començar a deteriorar-se. Van passar milers d’anys, milers de pluges torrencials, sequeres… fins i tot múltiples glaciacions. Tot el que n’ha quedat en l’actualitat és una dent mineralitzada, on qualsevol resta d’ADN ha estat esmicolada en una infinitat de fragments moleculars.
La resposta a la pregunta anterior és, doncs, que l’ADN no perdura eternament, es deteriora de forma gradual, es trenca i es transforma en altres molècules, i acaba esdevenint indetectable o ininterpretable per a nosaltres. Aquest deteriorament es veu accentuat en climes equatorials i tropicals, ja que les altes temperatures n’acceleren el procés. De fet, aquesta és una de les raons per les quals la gran majoria d’estudis d’ADN antic s’han focalitzat en mostres localitzades ben lluny dels tròpics. El «rècord mundial» de seqüenciació d’ADN antic es troba en 560.000-780.000 anys: un cavall preservat en permagel en les parts més septentrionals del Canadà, al territori de Yukon (Orlando et al., 2013). Fora d’aquestes situacions excepcionals per a la preservació, l’ADN normalment es pot obtenir d’organismes que van morir fa unes desenes de milers d’anys. Per tant, malgrat el seu gran potencial, l’ADN antic es veu limitat a obrir finestres al passat més recent.
«L’ADN no perdura eternament, es deteriora de forma gradual i esdevé indetectable o ininterpretable»
Hi ha cap esperança d’obtenir dades moleculars de Gigantopithecus de la cova de Chuifeng? La seva antiguitat suggereix que no gaires. Qualsevol traça del seu ADN es deteriora pel pas del temps i acaba completament malmesa pel clima humit i càlid del sud de la Xina. Tanmateix, les cèl·lules contenen altres tipus de molècules a banda de l’ADN. La majoria de llibres de biologia dediquen unes pàgines a l’anomenat «dogma central de la biologia»: l’ADN es transcriu a ARN i tot seguit aquest es tradueix a proteïnes. Malgrat que aquest model és una gran simplificació del flux real d’informació genètica dins una cèl·lula, obre un fil de llum per al nostre propòsit: seria possible seqüenciar l’ARN o les proteïnes d’una dent d’1,9 milions d’anys? L’ARN és molt semblant a l’ADN, una seqüència de nucleòtids on la timina (T) és reemplaçada per uracil (U); així doncs, la seva estabilitat i resistència al pas del temps és equiparable a la de l’ADN. Però… i les proteïnes?
«Les proteïnes semblen reunir totes les condicions necessàries per a estudiar el passat més llunyà, aquell que és inaccessible per l’ADN»
Les proteïnes són més estables que l’ADN. Ambdues molècules són cadenes de petites unitats (nucleòtids i aminoàcids, respectivament), però els enllaços entre les unitats de les proteïnes, els enllaços peptídics, són més resistents. A més a més, les proteïnes es pleguen en estructures tridimensionals que poden protegir-les del seu deteriorament (en certa manera, es troben plegades sobre si mateixes, fora de l’abast d’influències externes). Per últim, però no per això menys important, les proteïnes són més abundants; excloent-ne l’aigua, aproximadament el 3 % de la massa d’una cèl·lula és ADN, ben poc comparat amb el 50 % de la massa constituïda per proteïnes. Per tant, les proteïnes semblen reunir totes les condicions necessàries per a estudiar el passat més llunyà, aquell que és inaccessible per l’ADN. Tot i semblar una idea revolucionària, als anys cinquanta alguns investigadors ja havien documentat la detecció d’aminoàcids en fòssils. Malgrat això, la tecnologia per a seqüenciar proteïnes antigues no va existir fins als anys 2000, quan la comunitat científica va adoptar l’espectrometria de masses, una tècnica típicament utilitzada en disciplines de la física. Un espectròmetre de masses detecta la presència de diferents pèptids (fragments curts d’una proteïna) en una mostra, i ho aconsegueix analitzant-ne la massa (els «pesa»). La informació dels pèptids pot ser integrada a posteriori en seqüències de proteïnes de longitud més elevada.
El que ens pot dir una dent
A finals del 2019, un equip internacional va intentar obtenir proteïnes de l’esmalt del Gigantopithecus de Chuifeng mitjançant l’espectrometria de masses. L’intent va ser un èxit (Welker et al., 2019). Tenint en compte «l’edat termal» de la mostra (1,9 milions d’anys ajustats a la temperatura anual de la cova), són les dades moleculars de mamífer més antigues mai obtingudes. L’esmalt del Gigantopithecus és ric en sis proteïnes diferents. Mitjançant la comparació de les seqüències de proteïna obtinguda amb les de la resta de grans simis actuals (ximpanzés, bonobos, orangutans i humans), s’ha pogut establir que Gigantopithecus era un parent proper dels orangutans. D’altra banda, utilitzant el rellotge molecular (ritme al qual apareixen canvis en la seqüència d’una proteïna), es creu que els orangutans i Gigantopithecus van compartir un ancestre comú fa uns deu o dotze milions d’anys. A més, la detecció d’una proteïna gens comuna en l’esmalt d’altres simis permet hipotetitzar sobre les raons que hi ha rere la distintiva morfologia dental de Gigantopithecus. Si Gustav von Koenigswald estigués viu, de ben segur que quedaria bocabadat de veure com la tecnologia ha transformat la biologia en menys de cent anys.
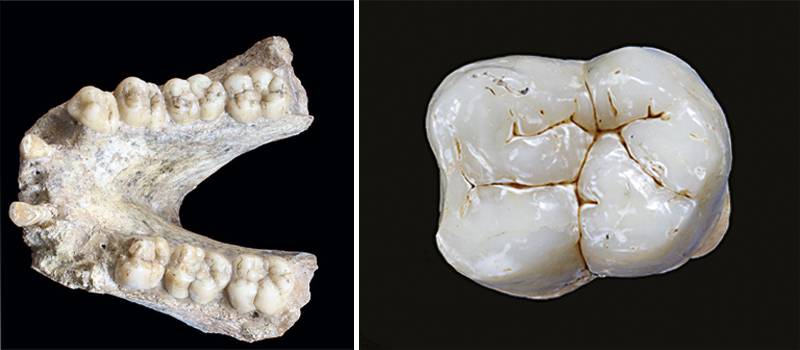
L’anàlisi de la mandíbula de Gigantopithecus blacki ha permès detectar una proteïna gens comuna en l’esmalt d’altres simis, el que permet hipotetitzar sobre les raons que hi ha rere la distintiva morfologia dental d’aquesta espècie. Aquest estudi, més enllà de les dades que s’han pogut conèixer del gran primat, obre un ventall enorme de noves oportunitats per a entendre el nostre passat. A la dreta, molar inferior de Gigantopithecus blacki trobat a la cova xinesa de Chuifeng. D’aquest esmalt s’han pogut extraure dades moleculars que fins al moment són les més antigues mai obtingudes d’un mamífer. / Wei Wang; Photo retouching: Theis Jensen.
Al marge d’ampliar el nostre coneixement sobre Gigantopithecus, l’estudi és important per raons no directament relacionades amb el primat gegant. Demostra que és possible obtenir dades moleculars d’organismes de milions d’anys d’antiguitat en climes subtropicals. Obre un ventall enorme de noves oportunitats per a entendre el nostre passat. Un bon exemple d’aquestes aplicacions també el trobem el 2019. Els denissovans són un grup d’homínids misteriós. Fins fa poc només s’havien pogut identificar a través de cinc fòssils trobats a la cova de Deníssova (origen del seu nom) a les muntanyes d’Altai, a Rússia. L’any 2010 es va obtenir la seqüència completa del seu genoma (tota la seqüència d’ADN d’un organisme) a partir de la falange d’una nena denissovana (Reich et al., 2010). Malgrat tot això, mai s’havia trobat un fòssil de Deníssova fora de les muntanyes d’Altai. Al maig del 2019, un equip d’investigadors va identificar una mandíbula de 160.000 anys d’antiguitat trobada a l’altiplà del Tibet com a denissovana mitjançant la paleoproteòmica (Chen et al., 2019). L’antiguitat i l’estat de conservació del fòssil feien l’extracció d’ADN antic impossible, i sense la paleoproteòmica, probablement mai hauríem après que la mandíbula pertanyia a una persona denissovana.
«La paleoproteòmica té el potencial d’omplir els buits que encara avui hi ha en l’arbre filogenètic dels humans i els seus ancestres»
D’altra banda, els casos especialment interessants poden ser aquells per als quals no disposem de dades moleculars. Qui eren els misteriosos «hòbbits» de l’illa de Flores (Homo floresiensis)? Era Homo heidelbergensis el nostre ancestre, o més aviat l’ancestre dels neandertals? I Homo erectus? Què podem aprendre d’un altre llinatge descobert recentment com Homo naledi? La llista podria seguir i seguir. L’aplicació de la paleoproteòmica té el potencial d’omplir els buits que encara avui hi ha en l’arbre filogenètic dels humans i els seus ancestres quan ens endinsem més enllà dels 500.000 anys en el passat.
Malgrat aquesta última oda al progrés, el nou camp de la paleoproteòmica té certes limitacions. Encara que les proteïnes contenen codificada informació genètica (tal com descriu el «dogma central de la biologia»), són menys informatives que l’ADN. Principalment per dues raons. Primer de tot, perquè part de la informació genètica es perd en la traducció d’ARN a proteïna. El codi genètic és redundant, és a dir, que hi ha diferents triplets d’ARN que codifiquen per a un mateix aminoàcid. Per exemple, l’aminoàcid serina està codificat per dos triplets de nucleòtids d’ARN: AGU i AGC. Així doncs, si trobem una serina en la seqüència d’una proteïna, no tenim cap eina per a discernir quin era el triplet original en l’ADN. En segon lloc, les proteïnes són fonamentals per al funcionament d’una cèl·lula, per tant, les regions d’ADN que codifiquen per proteïnes tenen una pressió selectiva molt gran, és a dir, els canvis de seqüència estan molt restringits, ja que qualsevol modificació podria implicar una pèrdua de funció. A efectes pràctics, una pressió selectiva alta resulta en una baixa diversitat de seqüència. Per exemple, el col·lagen és una proteïna que pràcticament té la mateixa seqüència en un humà que en un macaco. Aquesta observació s’explica per la gran importància del col·lagen: milions d’anys d’evolució han generat una seqüència òptima de la proteïna, i qualsevol mutació serà eliminada per l’efecte de la selecció natural. Per tant, obtenir dades moleculars de proteïnes pot resultar en seqüències extremadament similars entre organismes, fet que dificulta la reconstrucció de filogènies i establir relacions evolutives. Un exemple: imagineu que aconseguiu la seqüència del col·lagen d’una mostra d’Homo floresiensis. Després de dedicar-hi moltíssim temps i recursos, us adoneu que la seva proteïna i la nostra (Homo sapiens) són idèntiques, i que, per tant, no podeu aprendre gairebé res sobre Homo floresiensis. En resum, les proteïnes poden arribar a estar tan conservades en l’evolució que obtenir-ne la seqüència pot resultar en un exercici poc profitós.
La proteòmica també té altres reptes, en aquest cas relacionats amb les persones i el modus operandi de la comunitat científica. Ha d’anar amb compte de no caure en el mateix parany que l’ADN antic fa vint anys. Durant els anys noranta i principi dels 2000, certes publicacions van defensar haver obtingut ADN de dinosaures o insectes atrapats en ambre (al més pur estil Jurassic Park), troballes que en retrospectiva són difícils de creure. Més tard es va demostrar que aquests resultats eren producte de contaminació amb ADN modern o bé d’altres errors metodològics. La paleoproteòmica és un camp fascinant que acapararà molta atenció els propers anys. Així doncs, la comunitat científica haurà establir una llista de bones pràctiques, reproductibilitat i transparència si vol evitar caure en els mateixos errors del passat.
Sense cap mena de dubte, ens trobem en una etapa engrescadora per a la biologia evolutiva. La paleoproteòmica té el potencial per a explorar intervals de temps que eren del tot inaccessibles fins ara. Cal recordar que aquesta tècnica no es veu restringida a l’estudi de primats o humans, i pot influir en la recerca en altres animals, plantes, fongs, etc. L’any 2019 (com podeu veure, un any extremadament fructuós per a la paleoproteòmica i possiblement un auguri del que vindrà en el futur més proper), es va establir la posició filogenètica d’un rinoceront que va viure fa 1,7 milions anys (Cappellini et al., 2019). Només uns anys abans, una altra recerca va obtenir proteïnes de la closca d’un ou d’estruç de 3,8 milions d’anys a Laetoli (Demarchi et al., 2016), el mateix indret de les famoses petjades d’Australopithecus afarensis. Així doncs, molt probablement som a les beceroles d’una onada de troballes, impulsada per noves generacions de científics i científiques que potser canviaran per sempre més la nostra manera d’entendre la biologia evolutiva.
Referències
Cappellini, E., Welker, F., Pandolfi, L., Ramos-Madrigal, J., Samodova, D., Rüther, P. L., ... Willerslev, E. (2019). Early Pleistocene enamel proteome from Dmanisi resolves Stephanorhinus phylogeny. Nature, 574, 103–107. doi: 10.1038/s41586-019-1555-y
Chen, F., Welker, F., Shen, C.-C. , Bailey, S. E., Bergmann, I., Davis, S., ... Hublin, J.-J. (2019). A late Middle Pleistocene Denisovan mandible from the Tibetan Plateau. Nature, 569, 409–412. doi: 10.1038/s41586-019-1139-x
Demarchi, B., Hall, S., Roncal-Herrero, T., Freeman, C. L., Woolley, J., Crisp, M. K., ... Collins, M. J. (2016). Protein sequences bound to mineral surfaces persist into deep time. eLife, 5, e17092. doi: 10.7554/eLife.17092
Orlando, L., Ginolhac, A., Zhang, G., Froese, D., Albrechtsen, A., Stiller, M., ... Willerslev, E. (2013). Recalibrating Equus evolution using the genome sequence of an early Middle Pleistocene horse. Nature, 499, 74–78. doi: 10.1038/nature12323
Prüfer, K., Racimo, F., Patterson, N., Jay, F., Sankararaman, S., Sawyer, S., ... Pääbo, S. (2014). The complete genome sequence of a Neanderthal from the Altai Mountains. Nature, 505, 43–49. doi: 10.1038/nature12886
Reich, D., Green, R. E., Kircher, M., Krause, J., Patterson, N., Durand, E. Y., ... Pääbo, S. (2010). Genetic history of an archaic hominin group from Denisova Cave in Siberia. Nature, 468, 1053–1060. doi: 10.1038/nature09710
Welker, F., Ramos-Madrigal, J., Kuhlwilm, M., Liao, W., Gutenbrunner, P., De Manuel, M., ... Cappellini, E. (2019). Enamel proteome shows that Gigantopithecus was an early diverging pongine. Nature, 576, 262–265. doi: 10.1038/s41586-019-1728-8
Zhang, Y., & Harrison, T. (2017). Gigantopithecus blacki: A giant ape from the Pleistocene of Asia revisited. American Journal of Physical Anthropology, 162(S63), 153–177. doi: 10.1002/ajpa.23150