|
Evolution and disease: What made Salmonella pathogenic? The versatility and frequency with which genetic interchange takes place between bacteria constitute, to a great extent, the basis of their evolutionary success. Here we examine to what extent the systematic horizontal interchange is responsible for the evolution of pathogenicity in Salmonella. Moreover, knowing what other bacterial species give Salmonella its pathogenic character can be held up as a typical example of how studying evolution can be an effective procedure in the treatment of illnesses caused by microorganisms. La salmonel·la és un bacteri responsable de diverses malalties que afecten els humans i els animals domèstics. Aquests bacteris constitueixen una gran amenaça per a la nostra salut i benestar, i són responsables d’uns 16 milions de casos de salmonel·losi (incloent-hi febres tifoides) i més de 500.000 morts cada any. Fins fa poc de temps el gènere Salmonella, que consta de més de 2.000 espècies, es diferenciava en funció dels seus serotips, els seus orígens geogràfics o els animals que li fan d’hostes. Els serotips han estat tradicionalment definits en funció dels seus antígens flagel·lars i lipopolisacàrids, que són molècules que es troben en la superfície de la cèl·lula bacteriana. Això no obstant, no és possible deduir les vertaderes relacions genètiques entre les soques de bacteris a partir de les classificacions i de les dades serològiques. Basant-se en mètodes que poden resoldre les relacions genètiques entre llinatges, la majoria d’aquests 2.000 serotips de Salmonella s’han reclassificat en una única espècie denominada Salmonella enterica. A més a més, la majoria dels llinatges de la Salmonella que són patògens per als humans pertanyen a solament una de les set subespècies en què se subdivideix l’espècie S. enterica. Aquesta subespècie, que inclou el Typhi, un serotip que provoca febre tifoide en humans, també inclou el Typhimurium, que produeix gastroenteritis en humans i una malaltia similar al tifus en ratolins. Les estretes relacions de parentiu entre soques que causen malalties en distints hostes mamífers han portat a estudiar intensament els mecanismes patogènics del Typhimurium en ratolins com a model per a comprendre les malalties en humans. Pel que fa als humans, la Salmonella enterica i l’Escherichia coli són organismes molt diferents. L’E. coliSalmonella (vegeu figura 1), però, a diferència d’aquesta, l’E. coli és un resident inofensiu a la flora intestinal dels mamífers, incloent-hi els humans i s’utilitza com a eina en diferents estudis de genètica (figura 2). Això no obstant, d’acord amb un gran nombre de criteris moleculars i genètics, aquestes dues espècies de bacteris són molt similars. Per exemple, els cromosomes de l’Escherichia coli i del patogen Typhimurium són de la mateixa grandària, i tots dos comparteixen al voltant del 90% dels gens. Considerant la gran similitud genètica, quin és l’origen de les diferències que fan de l’E. coli un comensal inofensiu i de Salmonella un de patogen? L’antecessor comú de l’E. coli i de la Salmonella S’ha estimat que l’E. coli i la Salmonella van divergir a partir d’un antecessor comú fa al voltant de 100 milions d’anys, coincidint amb l’origen dels mamífers. Per tant, és probable que alguna de les característiques úniques dels mamífers, com ara l’homeotèrmia, l’ambient intestinal mamífer, i fins i tot la disponibilitat del sucre de la llet (lactosa), proporcionaren un nou nínxol ecològic que va permetre la divergència entre la Salmonella i l’E. coli. Quines van ser les característiques de l’organisme ancestral que va donar origen a aquestes dues subespècies de bacteri? Encara que és difícil reconstruir molts dels esdeveniments que van ocórrer en el passat llunyà, basant-se en consideracions ecològiques i genètiques, podem deduir que l’antecessor comú de l’E. coli i de la Salmonella va ser molt probablement un organisme comensal dels rèptils. Això significaria que la Salmonella d’alguna manera va evolucionar fins esdevenir un patogen i se suposa que l’E. coli va passar de ser un organisme patogen a un comensal. Sembla que l’E. coli, com també els seus antecessors, està predisposat a convertir-se en patogen. En consonància amb la noció segons la qual la Salmonella es va originar a partir d’un organisme comensal, un gran nombre de gens (al voltant de 50, segons un recompte recent) que es requereixen per a la virulència de la Salmonella també són presents en l‘E. coli. Aquests gens codifiquen enzims responsables de la biosíntesi de nutrients que són escassos dins dels teixits de l’hoste, factors reguladors transcripcionals i post-transcripcionals, proteïnes necessàries per a reparar danys en el DNA, i productes necessaris per a defensar-se dels antibiòtics produïts pels seus hostes; resumint, aquells gens que es requereixen per a interactuar amb un organisme hoste i sobreviure en el seu interior. Què fa que la Salmonella siga patògena Normalment, la Salmonella s’adquireix a través del consum d’aigua o menjar contaminat i ha de resistir el baix pH de l’estómac abans d’adherir-se a les cèl·lules de l’epiteli intestinal i penetrar en el seu interior. Aquells microorganismes invasors que causen malalties sistèmiques també han de ser capaços de sobreviure en la sang i de replicar-se en els macròfags del fetge i de la melsa. I aquells microorganismes que causen infeccions cròniques moltes vegades s’amaguen en la vesícula biliar dels individus infectats. Atès aquest únic i complex cicle de vida, ens plantegem que, a més del gran nombre de gens que comparteixen l’E. coli i la Salmonella, hauria d’haver-hi gens específics de Salmonella responsables de les interaccions particulars que es donen entre aquest patogen i els seus hostes. Així, fa pocs anys, vam començar a examinar el paper en la virulència dels gens específics de Salmonella. Investigant la funció de les regions del cromosoma específiques de la Salmonella, vam descobrir diversos gens que li confereixen l’habilitat d’envair –i de sobreviure-hi dins– les cèl·lules de l’hoste (habilitats que són fonamentals per al cicle de vida de la Salmonella en els animals infectats). Ara sabem que aquests gens són part d’un grup (cluster) molt gran de gens que és present en la Salmonella però no en l’E. coli. Aquests grups de gens virulents que són presents en els patògens, però absents dels bacteris benignes íntimament emparentats, se’ls ha denominat “illes de patogenicitat”. Nosaltres hem estudiat la seqüència de DNA completa i les funcions de molts dels gens que constitueixen una d’aquestes illes, concretament la denominada “illa de la patogenicitat de Salmonella 1″ (SPI-1). Quin és l’origen dels gens que solament són presents en una espècie? La transferència horitzontal de gens, és a dir, l’adquisició de gens procedents d’espècies llunyanament emparentades, ofereix a l’organisme receptor una forma molt ràpida d’adquirir noves funcions. L’illa de patogenicitat que hem estudiat posseeix diverses característiques que indiquen que la Salmonella la va adquirir per transferència horitzontal de gens. En primer lloc, aquesta illa de patogenicitat (SPI-1) posseeix una proporció de les bases nitrogenades guanina i citosina (42% G+C) diferent de la resta del genoma de la Salmonella (52% G+C). Com que el contingut en aquestes bases és bastant homogeni al llarg del cromosoma bacterià, regions amb proporcions de bases atípiques denoten que han estat adquirides per transferència horitzontal des d’un organisme que té una composició de bases diferent. En segon lloc, les dimensions, ordre i orientació dels gens dins d’aquesta illa són molt similars als gens d’invasió continguts en el plasmidi de la virulència d’un altre bacteri que causa disenteria (gènere Shigella). Com que els plasmidis són fragments de DNA que es transmeten fàcilment entre bacteris, això suggereix que els gens de la virulència que es troben en plàsmids es poden moure i transferir entre diferents espècies de bacteris patògens. Finalment, gens homòlegs d’aquests gens de la virulència són presents en un ampli rang de bacteris patògens que infecten plantes i animals. Seqüències estructuralment i funcionalment similars s’han detectat en bacteris dels gèneres Salmonella, Shigella, Yersinia, Erwinia, Xanthomonas i Pseudomonas (una distribució filogenètica que abona l’adquisició independent d’aquests gens per cada taxó). Múltiples illes de patogenicitat en la Salmonella L’adquisició de gens de virulència per la Salmonella no va ser un fet únic i aïllat. Investigacions realitzades en diversos laboratoris d’arreu el món han descobert que en aquesta espècie hi ha almenys cinc illes de patogenicitat i diversos petits “illots de patogenicitat”, cadascun dels quals contribueix a les extraordinàries característiques de virulència d’aquest microorganisme. Reconstruint la història evolutiva d’aquests gens podem establir que la majoria es van adquirir molt aviat en l’evolució de S. enterica, després de la seua separació d’E. coli. Mentre que E. coli va adquirir gens que li permeteren convertir-se en un comensal de l’intestí dels mamífers, com els que li permeten aprofitar el sucre de la llet, la Salmonella va adquirir uns altres gens que li facilitaren explotar els seus hostes mamífers de manera molt diferent. Però tots dos casos demostren que l’adquisició de gens per transferència horitzontal pot tenir una gran importància en l’evolució d’un organisme i ha canviat el caràcter ecològic i patogènic d’aquests bacteris. Howard Ochman. Dep. d’Ecologia i Biologia Evolutiva, Universitat d’Arizona. |
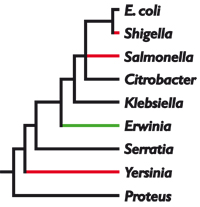
Figura 1: relacions de parentesc entre diferents gèneres de bacteris. En color roig els gèneres de bacteris patògens. «Aquests bacteris constitueixen una gran amenaça per a la nostra salut i benestar, i són responsables d’uns 16 milions de casos de salmonel·losi i més de 500.000 morts cada any»
|