L’ADN reescriu la nostra memòria
La recuperació dels desapareguts a través del seu perfil genètic

Els progressos continus en l’anàlisi de l’ADN amb finalitats forenses han anat marcant fites en la cerca de la identitat genètica en casos de caràcter criminal, de catàstrofes i de desaparicions. Els avanços tecnològics en l’estudi del genoma ens permeten inferir qui i com era l’amo d’unes restes trobades, per exemple, en una fossa comuna o una tomba anònima: on va viure, com era físicament, o quin era el seu origen familiar. L’anàlisi d’ADN d’interès forense aconsegueix, gràcies a una sèrie de variacions fixes entre individus, identificar-los individualment mitjançant el seu perfil genètic. Aquesta identificació es pot realitzar per comparació dels perfils de la resta humana amb perfils coneguts o per la seua compatibilitat amb els heretats dels seus familiars.
Paraules clau: persones desaparegudes, perfil genètic, perfil de metilació, perfil fenotípic, perfil biogeogràfic.
Recuperant el passat
Des del descobriment de la petjada genètica pel grup d’Alec Jeffreys, de la Universitat de Leicester, en 1985, s’han succeït progressos continus en l’anàlisi de l’ADN amb finalitats forenses, tant en el terreny tecnològic com en el tipus de marcadors genètics analitzats i en l’estandardització d’aquests. Gràcies a l’esforç de molts grups d’investigació i de treball de la Societat Internacional de Genètica Forense (Bär et al., 1997; Bodner et al., 2016; Carracedo et al., 2000; Mundorff, Bartelink i Mar-Cash, 2009; Parson et al., 2016), aquests avanços han anat marcant fites en la cerca de la identitat genètica per a l’esclariment de causes criminals, la gestió de grans catàstrofes (Prinz et al., 2007) o la investigació de desaparicions.
«Els avanços tecnològics en l’estudi del genoma ens permeten inferir qui i com era l’amo d’unes determinades restes»
L’ADN no es pot veure fàcilment, per això ha passat desapercebut durant tants anys, però pot llegir-se sempre, i cada vegada amb més claredat, en aquelles restes humanes oblidades per la transmissió familiar i cultural, la història de les quals podrem recordar o completar a través de la seua petjada genètica. Els avanços tecnològics en l’estudi del genoma ens permeten inferir qui i com era la persona a qui pertanyen unes determinades restes: on va viure, com era físicament, o quina era la seua genealogia (Figura 1). I amb l’avanç de la genòmica podrem deduir molts aspectes més de la identitat social que, ara com ara, només podem concebre amb la imaginació.
Sabem que no hi ha història sense memòria: per això sempre hi ha constància de l’èxit i la victòria i, en canvi, hem d’esforçar-nos per a recompondre el fracàs i la vergonya de la nostra espècie. No obstant això, si ja som capaços de llegir la nostra prehistòria genòmica neandertal (Green et al., 2010; Noonan et al., 2006) i d’altres ancestres extingits fa milers d’anys, una prehistòria larvada en les línies del nostre propi ADN (Dannemann i Kelso, 2017), prompte serem capaços de recuperar la memòria d’aquelles persones les restes de les quals es van oblidar o es van ocultar en el segle passat, fora en període de guerra o de postguerra. De la mateixa manera, prompte serem capaços de rescatar els qui són víctimes de l’oblit en el present, víctimes dels nombrosos conflictes bèl·lics actuals i de les onades de migrants que aquests generen, a més de la fam, la discriminació i la injustícia institucionalitzada.
La tecnologia continua avançant, millorant el maneig dels materials que hem d’estudiar en la identificació de les restes d’éssers estimats exhumats de fosses comunes o de tombes anònimes, o d’aquells recuperats de les platges del Mediterrani. D’igual manera, cada dia milloren els mètodes d’extracció, restauració i lectura de la seqüència genòmica. Així, no solament podem fer anàlisis de parentiu o comparar els resultats amb mostres indubtables d’una víctima, sinó que fins i tot som capaços, a vegades, d’obtenir alguns dels seus trets fenotípics o informació sobre l’origen dels seus ancestres, i establir una aproximació probabilística de la seua edat o d’algunes característiques del seu estil de vida. No obstant això, no voldríem fer l’efecte que és molt senzill establir la identitat d’unes restes humanes. Retornar-los el nom i el cognom a partir de la informació indirecta aportada per l’estudi científic no sempre és possible, ja que moltes vegades estan tan degradades que no poden explicar-nos el que esperem conèixer: la seua identitat individualitzada.
En els murs d’una ciutat es podia llegir «sense memòria no hi ha aprenentatge»: qui fa memòria aposta pel futur, per conservar la seua identitat i el seu rigor, i això li permetrà sobreviure a la famosa pàtina del temps. Sense memòria no podem atresorar coneixement, ni aprendre dels encerts i errors d’èpoques passades, ja siga a través de les seues petjades o de les seues restes, que ens narren la història del que va succeir i que no coneixem. La bona història ha de fer-se com una suma de les veritats: les dels herois i els pròcers, però també les dels testimonis anònims o ocults. A aquests, cal donar-los una identitat, perquè puguem aprendre sobre el moment en què van viure, van somiar o van desaparèixer. En aquesta labor s’afanyen historiadors, artistes, paleontòlegs, antropòlegs i, des de fa unes poques dècades, els genetistes.
Després d’aquestes reflexions intentarem explicar en aquest text, gràcies als avanços en la tecnologia, quines preguntes podem fer a aquestes restes, de quines preguntes obtindrem una ràpida resposta i per a quines caldrà esperar encara.
Mirant el present
Des de la seqüenciació completa del genoma humà a començament del segle XXI (Venter et al., 2001) fins avui, s’han desenvolupat múltiples tècniques que permeten millorar l’anàlisi d’ADN d’interès forense, utilitzat per a col·laborar en la identificació de les restes de desapareguts, tant en conflictes armats com en catàstrofes o tràfic de persones.
Aquesta «tipificació» d’ADN s’aconsegueix gràcies a l’esclariment de la variació genètica entre humans: el nostre genoma presenta una sèrie de variacions fixes entre subjectes que permeten identificar-los individualment. Aquesta identificació pot realitzar-se per comparació dels perfils de les restes humanes amb perfils coneguts o per la seua compatibilitat amb l’herència de progenitors identificats, com ocorre quan realitzem la investigació forense més bàsica: la investigació biològica de la paternitat.
En aquest cas, comparem el perfil genètic del fill amb el de la seua mare (de la qual usualment no es dubta, ja que existeix documentació del part). La variació genètica entre els dos genomes, és a dir, allò que no ha heretat el fill de la mare biològica, obligatòriament ha de provenir del progenitor masculí o pare biològic. Aquest tipus de mètode també s’aplica en els casos de desapareguts, en els quals compararem la informació genètica dels familiars més idonis amb la del desaparegut (ja siga viu o a partir de les seues restes), confrontant l’herència d’aquesta informació de manera «neutra» –en aquesta circumstància, dubtem dels dos pares–, ja que no tenim la certesa que aquestes restes siguen o no del familiar desaparegut.

Figura 2. Un perfil genètic de marcadors tipus STR és com un codi de barres propi de cada persona. En la imatge observem un perfil d’aquest tipus obtingut mitjançant anàlisi de fragments d’ADN d’un individu anònim en un seqüenciador automàtic. Els colors corresponen a cadascun dels marcadors fluorescents usats i cada pic de la imatge de l’electroferograma representa una variant (al·lel) de l’individu analitzat. Per a elaborar aquest perfil, s’ha emprat un equip comercial per a analitzar 17 microsatèl·lits en diferents cromosomes i inclou el gen de l’amelogenina (el primer pic de la fila en blau), que permet conèixer el sexe masculí (XY) o, com és el cas d’aquest perfil, femení (XX). / Mercedes Aler
Aquest tipus d’identificació es realitza tècnicament analitzant petits fragments del genoma, els microsatèl·lits o STR (de les sigles en anglès de short tandem repeats), que donaran un codi o perfil genètic concret. Es tracta de repeticions de grups de nucleòtids (en general tetra- o pentanucleòtids) que generen fragments d’ADN amb diferents longituds entre ells i molt polimòrfics –és a dir, molt variables entre les persones– i que, quan s’analitzen de manera automàtica en un seqüenciador, ens proporcionen el perfil genètic, una espècie de codi de barres únic i exclusiu de cada persona (Figura 2), excepte per a bessons idèntics (univitelins), perquè en aquest cas tots dos posseeixen el mateix perfil.
Però quan no podem obtenir aquest perfil complet per una mala qualitat de la mostra de partida (restes deteriorades amb ADN molt fragmentat o degradat) o necessitem més respostes, podem ampliar l’estudi amb un altre tipus de polimorfismes molt més abundants en el nostre genoma com ara els SNP (sigles en anglès de single nucleotide polymorphism, “polimorfismes de nucleòtid únic”) i els indel (mutacions per inserció o deleció de nucleòtids). Analitzant-los augmentem la informació necessària per a resoldre moltes de les preguntes que no tenien resposta amb els STR.
«El perfil genètic és una espècie de codi de barres únic i exclusiu de cada persona excepte per a bessons idèntics»
Hi ha milions d’aquests SNP en diferents segments del genoma, i com que són fragments d’ADN molt més petits que els STR, és més difícil que es degraden i fragmenten. Aquesta anàlisi, tot i ser molt útil, necessita protocols de treball que exigeixen més temps de processament i, encara que la taxa de mutació és menor que la d’un STR, es necessita estudiar més SNP per a obtenir la mateixa informació que amb els STR (Gill, 2001), ja que són menys polimòrfics.
Els indel, considerats també com un tipus de SNP, consisteixen en polimorfismes bial·lèlics simples d’inserció i deleció d’un o pocs parells de nucleòtids. Són senzills d’analitzar i poden estudiar-se per separació de grandàries –mitjançant l’anàlisi de fragments–, com en el cas dels STR. Tots aquests polimorfismes s’analitzen amb mètodes semblants, que comporten l’amplificació de múltiples fragments alhora (reacció múltiple), que després se separen en un seqüenciador automàtic, gràcies al fet que els hem marcats amb un producte fluorescent durant la reacció en cadena de la polimerasa (o PCR en les sigles en anglès de polymerase chain reaction).
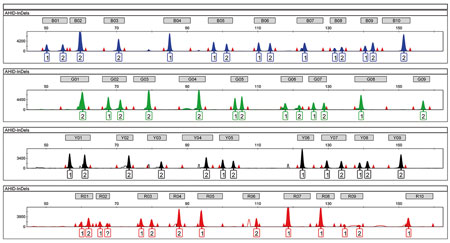
Figura 3. Quan la mala qualitat d’una mostra d’ADN dificulta l’elaboració d’un perfil genètic complet, l’estudi pot ampliar-se analitzant altres polimorfismes abundants en el nostre genoma, com els indel, polimorfismes bial·lèlics simples d’inserció i deleció d’un o pocs parells de nucleòtids. En la imatge, podem observar un perfil genètic de 38 marcadors autosòmics de tipus indel per a identificació, estudiats mitjançant anàlisi de fragments d’ADN en un seqüenciador automàtic. Cada pic de la imatge de l’electroferograma representa una variant (al·lel) de l’individu analitzat. / Mercedes Aler
De l’anàlisi de tots es pot obtenir un perfil genètic específic per a cada individu, amb diferent nombre de llocs del genoma analitzats: uns vint llocs diferents del genoma per a microsatèl·lits o STR, uns cinquanta per a un perfil de SNP i uns quaranta per a un d’indel (Figura 3). El nombre de polimorfismes analitzats, del qual s’obté el perfil genètic, varia segons el producte utilitzat per a l’anàlisi: els equips comercials contenen entre disset i vint-i-quatre i els generats per grups d’investigació inclouen aquells que compleixen l’objectiu concret del seu projecte (per exemple, l’elaboració d’un perfil fenotípic o ancestral).
En els últims anys, l’anàlisi de variacions de tipus SNP en l’ADN relacionades amb característiques físiques visibles –l’anomenat FDP, per les sigles en anglès de forensic DNA phenotyping– va guanyant importància i, en aquest context, podria ser útil quan volem conèixer informació més específica del desaparegut per comparar-la amb la que tenim de referència proporcionada pels familiars o persones pròximes (una imatge fotogràfica o un document descriptiu de trets, com ara el color dels ulls, de la pell, la forma del cabell…). És a dir, encara que l’estudi de marcadors de trets fenotípics no ens serveix per si mateix per a identificar la persona desapareguda, aquests marcadors sí que aportaran respostes concretes sobre les característiques físiques que puguen ajudar a identificar-la. Així, si les restes que estudiem coincideixen amb l’esperat quant a l’aparença i genètica de la persona que busquem, estarem més prop de la resposta.
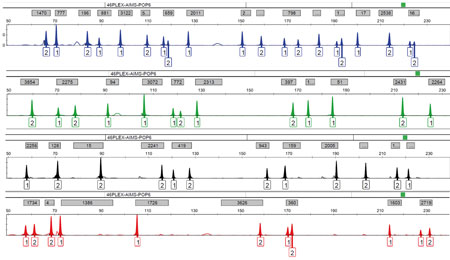
Figura 4. L’anàlisi dels SNP (polimorfismes nucleotídics simples, en les seues sigles en anglès) resulta molt útil, ja que, com que es tracta de fragments d’ADN molt petits, és més difícil que es degraden i fragmenten. Existeixen també els marcadors informatius d’ancestralitat (AIM en les seues sigles en anglès), per mitjà dels quals pot elaborar-se el perfil biogeogràfic d’un individu que ens servirà per a buscar persones desconegudes que no són identificables amb el perfil estàndard d’ADN. En la imatge, perfil genètic de 46 marcadors autosòmics d’ancestralitat de tipus indel estudiats mitjançant anàlisis de fragments d’ADN en un seqüenciador automàtic. Cada pic de la imatge de l’electroferograma representa una variant (al·lel) de l’individu analitzat. / Mercedes Aler
I, encara més, amb les variants dels SNP específics de població denominats AIM (de l’anglès ancestry informative markers, “marcadors informatius d’ancestralitat”) (Figura 4) podem predir, mitjançant l’aplicació de potents paràmetres probabilístics, l’anomenat «perfil biogeogràfic», que ens servirà per a buscar persones desconegudes que no són identificables amb el perfil estàndard d’ADN. Aquest conjunt de marcadors genètics permet predir l’ascendència a escala continental i fins i tot subcontinental, si bé cal tenir en compte que, en l’era de la globalització i de les migracions humanes ràpides en què vivim, aquest origen ancestral no té per què coincidir amb la nacionalitat (Lipphardt, Toom, Mupepele i Lemke, 2017).
Els marcadors AIM ja es fan servir en l’actualitat en la resolució de casos criminals en els quals, per exemple, el perfil genètic d’una taca d’esperma o de sang no ens permet fer la identificació si no tenim ja el perfil d’un sospitós per a comparar-los. En aquests casos de perfils «anònims», la predicció del perfil biogeogràfic i del fenotípic pot donar-nos respostes sobre, com ja hem esmentat, l’ascendència geogràfica (europea, asiàtica, africana…), a més del color dels ulls, del cabell i de la pell del donant de la taca, i proporcionar-nos d’aquesta manera informació de trets del sospitós anònim, la qual cosa permet avançar en la investigació policial. Això és el que va ocórrer en el cas del crim de la jove Eva Blanco en 1997, en què el perfil d’ADN autosòmic i el del cromosoma Y d’un dels investigats va suggerir que el germà d’aquest podria ser l’autor del crim, que es va poder resoldre així quasi vint anys després mitjançant l’ús d’aquests marcadors. A pesar d’aquest cas, aquest tipus d’investigacions basades en l’estudi dels AIM no poden realitzar-se de manera rutinària en els laboratoris forenses d’Espanya, perquè no estan a l’abast de tots els centres, ni estan regulades de manera específica en la nostra legislació. Quan es fan servir, sempre es fa sota el mandat de l’autoritat judicial.
«L’anomenat “perfil biogeogràfic” ens servirà per a buscar persones desconegudes que no són identificables amb el perfil estàndard d’ADN»
D’altra banda, la seqüenciació paral·lela massiva o de nova generació (NGS en les sigles en anglès de next generation sequencing) està revolucionant, igual com ocorre amb el diagnòstic genètic clínic, les aplicacions forenses de l’anàlisi de les variacions d’ADN. Aquesta tècnica, encara en desenvolupament per a la solució de casos forenses, ens permet analitzar molts i diferents polimorfismes alhora (STR, SNP, indel) al costat de la seqüència de totes les variants internes nucleotídiques dels STR, fet que augmenta el seu poder de discriminació. Però el major avantatge és la possibilitat d’obtenir bons resultats en material altament degradat perquè es poden analitzar fragments petits amb gran profunditat de lectura (és a dir, la tècnica permet realitzar múltiples lectures sobre la mateixa mostra, la suma de les quals permet aquesta precisió o profunditat). L’NGS facilita també l’anàlisi de marcadors de metilació –la petjada que deixa el pas del temps en l’ADN– per a la predicció de l’edat biològica (Abbott, 2018; Freire-Aradas et al., 2016).
Una altra aplicació de l’NGS és la distinció de bessons univitel·lins a partir de mostres biològiques. Aquest era un dels reptes de la medicina forense, ja que, en casos criminals, la presumpció d’innocència preval si no es pot demostrar quin dels dos bessons va cometre el delicte. A través de la seqüenciació de genomes complets a altes cobertures i detectant mutacions postcigòtiques, aquesta diferenciació ha deixat de ser un impossible (Vidaki i Kayser, 2017).
Finalment, cal esmentar, per la gran importància que té, l’ús de bases de dades poblacionals per a obtenir els resultats probabilístics, així com les bases de dades d’interès forense, regulades per llei i en les quals es «depositen» perfils genètics anònims, de convictes i de familiars de desapareguts, totes les quals amb interès de servir com a identificador si es donara el cas que foren útils (Ley orgánica 10/2007).
Esperant el futur
El futur predictible portarà avanços en l’epigenètica; sobretot en la cerca d’aplicacions a l’existència constatada de canvis moleculars en les cèl·lules deguts a l’exposició ambiental al llarg de la vida (Vidaki i Kayser, 2017). L’anàlisi d’aquestes variacions d’origen ambiental en el nostre genoma, ocorregudes per la metilació progressiva de l’ADN, permet obtenir –analitzant marcadors de tipus CpG– «perfils de metilació» d’interès forense específic en l’estudi de restes biològiques escasses i amb molt poc ADN, per a poder respondre les preguntes següents: De quin tipus de teixit és la resta (sang, semen, saliva…)? Quina edat biològica té el donant? A més, el perfil de metilació podria informar sobre l’estil de vida d’un individu: el consum de certes substàncies (tabac, alcohol o fàrmacs), l’activitat física, la dieta en les primeres fases de la vida, etc., perquè sabem que tot això influeix en l’estructura i aparença corporal, per la qual cosa, buscant i analitzant marcadors específics, es podrien establir aquests trets.
És evident, però, que l’ús d’aquestes variacions implica l’esclariment previ de patrons i processos complexos i variables que representen un repte a causa de la seua naturalesa quantitativa. També s’haurien de superar les barreres tecnològiques que comporta el fet de treballar amb mètodes de genotipificació múltiple, per a poder realitzar l’anàlisi qualitativa de diversos marcadors simultàniament. Caldrà identificar els marcadors epigenètics adequats, comprovar que s’obtenen resultats a partir d’una escassa quantitat d’ADN inicial, aprendre i comprendre l’abast d’aquests, desenvolupar models estadístics de predicció i, tan important com l’anterior, establir una supervisió i valoració ètica i social dels beneficis i riscos d’aquest tipus d’investigacions.
No voldríem acabar sense esmentar les possibilitats futures del microbioma (ADN dels microorganismes presents en un organisme), que podrà aportar des de dades per a la identificació personal fins a dades sobre els llocs on ha viscut l’individu i fins i tot els seus hàbits. És encara un camp d’estudi que està en el seu començament pel que fa a les aplicacions forenses, però de grans possibilitats no exemptes de dificultats tècniques i teòriques apassionants.
«Amb les tècniques de seqüenciació massiva entrem de ple en el coneixement de la intimitat genòmica i epigenòmica»
Finalment, hi ha plantejaments ètics i legals (Samuel i Prainsack, 2019) pendents sobre l’aplicació d’aquests avanços i dels futuribles, en raó al dret a la privacitat o al dret a no saber (Figura 5). Amb les tècniques de seqüenciació massiva entrem de ple en el coneixement de la intimitat genòmica i epigenòmica amb conseqüències possibles en el camp de la salut, del treball, de l’estil de vida i de l’exposició a determinats factors ambientals. A més, s’ha de tenir en compte la importància de l’avaluació i interpretació de les dades i de delimitar-les adequadament per a l’aplicació prevista, a fi d’evitar errors d’interpretació de resultats. Tal com assenyala la Comissió Internacional sobre Persones Desaparegudes (Parsons, Huel, Bajunović i Rizvić, 2019), aquests errors poden tenir conseqüències socials o judicials en les identificacions realitzades en zones de conflicte bèl·lic o després de grans catàstrofes.
Com ja hem comentat, tots aquests avanços exigeixen una regulació adequada, però, com sempre, la tecnologia avança més ràpidament que el dret. Així doncs, haurem de començar a legislar per a ajustar l’ús que en fem sense que això afecte els drets fonamentals de les persones.
Referències
Abbott, A. (2018). European scientists seek ‘epigenetic clock’ to determine age of refugees. Nature, 561(7721), 15. doi: 10.1038/d41586-018-06121-w
Bär, W., Brinkmann, B., Budowle, B., Carracedo, A., Gill, P., Lincoln, P., … Olaisen, B. (1997). DNA recommendations. Further report of the DNA Commission of the ISFH regarding the use of short tandem repeat systems. International Society for Forensic Haemogenetics, 110(4), 175–176.
Bodner, M., Bastisch, I., Butler, J. M., Fimmers, R., Gill, P., Gusmão, L., … Parson, W. (2016). Recommendations of the DNA Commission of the International Society for Forensic Genetics (ISFG) on quality control of autosomal Short Tandem Repeat allele frequency databasing (STRidER). Forensic Scientific International: Genetics, 24, 97–102. doi: 10.1016/j.fsigen.2016.06.008
Carracedo, A., Bär, W., Lincoln, P., Mayr, W., Morling, N., Olaisen, B., … Wilson M. (2000). DNA commission of the International Society for Forensic Genetics: Guidelines for mitochondrial DNA typing. Forensic Scientific International, 110(2), 79–85.
Dannemann, M., & Kelso, J. (2017). The contribution of Neanderthals to phenotypic variation in modern humans. American Journal of Human Genetics, 101(4), 578–589. doi: 10.1016/j.ajhg.2017.09.010
Freire-Aradas, A., Phillips, C., Mosquera-Miguel, A., Girón-Santamaría, L., Gómez-Tato, A., Casares de Cal, M., … Lareu, M. V. (2016). Development of a methylation marker set for forensic age estimation using analysis of public methylation data and the Agena Bioscience EpiTYPER system. Forensic Science International: Genetics, 24, 65–74. doi: 10.1016/j.fsigen.2016.06.005
Green, R. E., Krause, J., Briggs, A. W., Maricic, T., Stenzel, U., Kircher, M., … Pääbo, S. (2010). A draft sequence of the Neanderthal genome. Science, 328(5979), 710–722. doi: 10.1126/science.1188021
Gill, P. (2001). An assessment of the utility of single nucleotide polymorphisms (SNPs) for forensic purposes. International Journal of Legal Medicine, 114(4-5), 204–210.
Ley orgánica 10/2007, de 8 de octubre, reguladora de la base de datos policial sobre identificadores obtenidos a partir del ADN. (2007). Consultat en
https://www.boe.es/eli/es/lo/2007/10/08/10/con
Lipphardt, V., Toom, V., Mupepele, A.-C., & Lemke, T. (2017). Open letter on critical approaches to forensic DNA phenotyping and biogeographical ancestry. Consultat el 10 de desembre, de 2018, en http://www.fb03.uni-frankfurt.de/67048840/Lipphardt-et-al-2017---OPEN-LETTER-ON-CRITICAL-APPROACHES-TO-FORENSIC-DNA-PHENOTYPING-AND-BIOGEOGRAPHICAL-ANCESTRY.pdf
Mundorff, A. Z., Bartelink, E. J., & Mar-Cash, E. (2009). DNA preservation in skeletal elements from the World Trade Center disaster: Recommendations for mass fatality management. Journal of Forensic Sciences, 54(4), 739–745. doi: 10.1111/j.1556-4029.2009.01045.x
Noonan, J. P., Coop, G., Kudaravalli, S., Smith, D., Krause, J., Alessi, J., … Rubin, E. M. (2006). Sequencing and analysis of Neanderthal genomic DNA. Science, 314(5802), 1113–1118. doi: 10.1126/science.1131412
Parson, W., Ballard, D., Budowle, B., Butler, J. M., Gettings, K. B., Gill, P., … Phillips, C. (2016). Massively parallel sequencing of forensic STRs: Considerations of the DNA commission of the International Society for Forensic Genetics (ISFG) on minimal nomenclature requirements. Forensic Science International: Genetics, 22, 54–63. doi: 10.1016/j.fsigen.2016.01.009
Parsons, T. J., Huel, R. M. L., Bajunović, Z., & Rizvić, A. (2019). Large scale DNA identification: The ICMP experience. Forensic Science International: Genetics, 38, 236–244. doi: 10.1016/j.fsigen.2018.11.008
Prinz, M., Carracedo, A., Mayr, W. R., Morling, N., Parsons T. J., Sajantila A., … Schneider P. M. (2007). DNA Commission of the International Society for Forensic Genetics (ISFG): Recommendations regarding the role of forensic genetics for disaster victim identification (DVI). Forensic Science International: Genetics, 1(1), 3–12. doi: 10.1016/j.fsigen.2006.10.003
Samuel, G., & Prainsack, B. (2019). Forensic DNA phenotyping in Europe: Views «on the ground» from those who have a professional stake in the technology. New Genetics and Society, 38(2), 119–141. doi: 10.1080/14636778.2018.1549984
Venter, J. C., Adams, M. D., Myers, E. W., Li, P. W., Mural, R. J., Sutton, G. G., … Zhu, X. (2001). The sequence of the human genome. Science, 291(5507), 1304–1351. doi: 10.1126/science.1058040
Vidaki, A., & Kayser, M. (2017). From forensic epigenetics to forensic epigenomics: Broadening DNA investigative intelligence. Genome Biology, 18(1), 238. doi: 10.1186/s13059-017-1373-1