I la biologia esdevingué enginyeria
L’adopció d’estàndards per a sistemes vius
Durant dècades, els biòlegs moleculars han estat eliminant o inserint gens en tota mena d’organismes amb una intenció biotecnològica o simplement per a generar coneixement fonamental. La biologia sintètica fa un pas més enllà i incorpora marcs conceptuals procedents de la computació, l’electrònica i el disseny industrial. Aquest canvi permet plantejar la creació d’objectes biològics complexos que anteriorment es consideraven massa difícils d’assemblar. Per a això, cal adoptar les etapes de qualsevol procés de producció industrial: disseny, fabricació dels components, muntatge i manufactura final. Aquest objectiu fa necessari estandarditzar els formats físics i funcionals dels components implicats, els mètodes d’assemblatge de DNA, les mesures d’activitat i els llenguatges descriptius.
Paraules clau: biologia sintètica, estàndard, repressilador, repositori, ortogonalitat.
L’efecte fundacional: la biologia vista pels enginyers
Encara que la història de la biologia sintètica que coneixem es remunta molt de temps arrere, el naixement de la seua versió contemporània es pot situar clarament en l’Institut Tecnològic de Massachussetts (MIT, en les seues sigles en anglès) al començament dels anys 2000. En aquella època, Tom Knight, un professor d’intel·ligència artificial en el Departament de Ciències de la Computació del MIT, va començar a formalitzar la idea d’abordar els sistemes biològics utilitzant com a marc interpretatiu tota l’artilleria conceptual de l’enginyeria elèctrica, industrial i computacional. Per a això, calia adaptar a les entitats vives les abstraccions fonamentals dels enginyers quan analitzen i dissenyen objectes amb diferents graus de complexitat. D’aquesta manera, el conegut com a dogma central de la biologia molecular se substitueix per un marc interpretatiu altament abstracte en el qual les parts biològiques donen lloc als dispositius i aquests als mòduls i sistemes (Figura 1). I el context evolutiu per a explicar l’origen de les funcions biològiques s’aparca per a posar tot l’èmfasi en la lògica relacional que fa que els sistemes vius funcionen ací i ara (De Lorenzo, 2018).
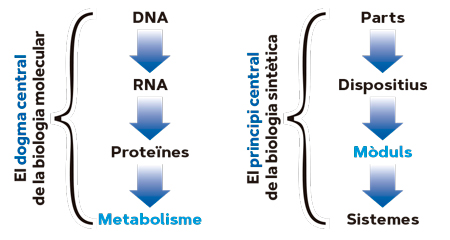
Figura 1. Del dogma central de la biologia molecular al marc conceptual de la biologia sintètica. En el primer cas, l’èmfasi és la transferència d’informació del DNA a les proteïnes i el metabolisme. En contrast, la biologia sintètica es preocupa de la lògica relacional i composicional dels sistemes vius i aplica abstraccions típiques de l’enginyeria als objectes biològics. / Font: De Lorenzo (2018)
Aquesta «opció hermenèutica» permet una visió dels sistemes vius compatible però molt diferent de la de la biologia molecular. Mentre que aquesta –fundada per científics atòmics de la postguerra mundial– va promoure una visió de les entitats biològiques a través dels ulls de la física, la biologia sintètica ambiciona reinterpretar-les a través de l’enginyeria (Andrianantoandro, Basu, Karig i Weiss, 2006). Un efecte col·lateral d’aquest nou punt de vista és que, a diferència de la biologia molecular, l’agenda de la biologia sintètica no té a veure només amb la comprensió dels objectes biològics existents, sinó també amb la seua modificació racional per a donar lloc a propietats i funcionalitats noves, en alguns casos, d’alt valor econòmic. En realitat, un dels grans horitzons de la biologia sintètica és fer rigorosament real la paraula enginyeria, que de manera metafòrica es va associar a genètica durant la revolució del DNA recombinant a la fi dels anys setanta del passat segle. Per contra, el discurs fundacional de la biologia sintètica declara que és possible passar de l’analogia a la metodologia i del bricolatge genètic de prova i error al plantejament racional de sistemes vius complexos, tal com un enginyer fa amb els seus dissenys tècnics (De Lorenzo, 2018; Endy, 2005). Per a això, primer cal desconstruir els sistemes biològics existents en un catàleg de parts que, una vegada estandarditzades, poden reconnectar-se amb una lògica predeterminada per a generar funcionalitats noves.
«La biologia sintètica ambiciona la reinterpretació de les entitats biològiques a través de l’enginyeria»
Del reeiximent d’aquest escenari dona compte l’extraordinari èxit de l’anomenat Repositori de Parts Biològiques Estandarditzades inicialment creat i depositat en el MIT i que va créixer en paral·lel a la competició internacional de màquines genètiques o iGEM, també promoguda en el seu moment pels teòrics de la biologia sintètica en la mateixa institució (Galdzicki, Rodriguez, Chandran, Sauro i Gennari, 2011). Aquesta competició, que arriba fins al dia d’avui, ha estat fonamental per a propagar la idea de la biologia-com-a-enginyeria a través de nombroses universitats a tot el món i ha estat en si mateixa un immens experiment educacional que ha iniciat diverses generacions de joves en els conceptes fonamentals de la biologia sintètica.
Però retrocedim un moment al principi. En què es basava l’optimisme manifestat en els començaments per Tom Knight i els seus col·legues del MIT? En els anys previs a la conceptualització de la biologia sintètica, van aparèixer tres articles que molts consideren fundacionals del camp, encara que en cap dels casos així s’expresse explícitament en el text de les publicacions corresponents. Un va ser la descripció l’any 2000 pel grup de M. Elowitz de l’anomenat repressilador: un circuit genètic entre tres repressors transcripcionals mútuament inhibidors que en certes condicions donen lloc a l’expressió cíclica d’un gen reporter (que, en aquest cas, codificava una proteïna fluorescent), que actua com a indicador visual del funcionament del sistema (Elowitz i Leibler, 2000; Figura 2A). L’angle revolucionari d’aquest treball era que aquest comportament estava dissenyat racionalment amb principis d’enginyeria i amb una configuració totalment artificial (no hi ha casos similars en el món natural). Encara així, el comportament responia fidelment a un model matemàtic simulable en un ordinador.
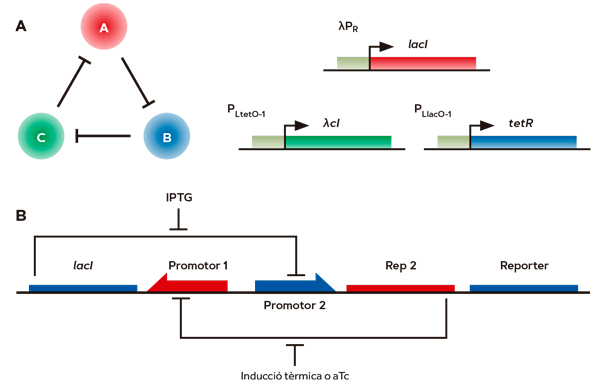
Figura 2. Dues de les construccions genètiques fundadores de la biologia sintètica. A) El repressilador. Aquest dispositiu genètic és un oscil·lador de l’expressió genètica in vivo que es construeix amb tres repressors, les proteïnes LacI, TetR, λCI (codificades pels gens lacI, tetR, λcI) i on cadascuna actua com a inhibidora de les següents a força de reprimir els promotors corresponents (λPR, PLlacO-1 y PLtetO-1), cadascun dels quals controla l’expressió del repressor següent. Aquest comportament estava dissenyat amb principis d’enginyeria i amb una configuració totalment artificial (no hi ha casos similars en el món natural). El comportament responia fidelment a un model matemàtic simulable en un ordinador. B) L’interruptor. Aquest sistema es basa en dos dels repressors transcripcionals usats en el repressilador, però organitzats de manera que s’inhibeixen mútuament (LacI inhibeix el promotor de tetR i viceversa). Això origina dos estats estables en els quals s’activa un promotor o s’activa l’altre, mai els dos alhora. Això es pot revertir amb l’addició d’un senyal inductor (per exemple, mitjançant l’ús del reactiu IPTG, o de l’anhidrotetraciclina o aTc), que desreprimeix un dels components. Al seu torn, això permet l’expressió de l’altre repressor i provoca un canvi d’estat del dispositiu. De nou, el sistema in vivo obeïa escrupolosament les regles imposades pel seu dissenyador humà i responia al model predictiu. / Font: Figura A: Elowitz i Leibler (2000), i Figura B: Gardner, Cantor i Collins (2000)
El segon article fundacional va ser publicat pel grup de J. Collins en el mateix any i descrivia el disseny i funcionament d’un interruptor genètic (toggle switch), assemblat amb dues de les parts biològiques (els repressors transcripcionals) utilitzades en el cas anterior (Gardner, Cantor i Collins, 2000; Figura 2B). Però, en aquest cas, cablades d’una manera molt diferent i que donava lloc a l’expressió estable de dos gens alternatius. De nou, el sistema in vivo obeïa escrupolosament les regles imposades pel seu dissenyador humà i responia al model predictiu. En una tercera publicació en l’any 2000 del laboratori de L. Serrano, es descrivien circuits genètics simples en els quals s’introduïen de manera racional llaços de retroalimentació negatius que donaven estabilitat al sistema (Becskei i Serrano, 2000). En cadascun dels tres casos el missatge era el següent: es poden aplicar principis d’enginyeria als sistemes vius, tant per a entendre com funcionen com per a fer que funcionen predictiblement d’una altra manera. L’impacte conceptual d’aquestes publicacions va ser enorme. I després de la corresponent formulació de la disciplina en el MIT per Knight i els seus col·laboradors Drew Endy (Endy, 2005) i Ron Weiss (Andrianantoandro et al., 2006) podem dir que la biologia sintètica havia nascut.
L’abecé de la biologia sintètica
La noció bàsica darrere de la biologia sintètica és que qualsevol sistema biològic pot considerar-se com una combinació complexa d’elements funcionals i independents, no gaire diferents als que es troben en els dispositius fets per l’home. Sobre aquesta premissa, els objectes corresponents es poden descriure com la combinació d’un nombre limitat de components i reconstruïts en una configuració diferent, bé per a modificar les propietats existents o per a aconseguir-ne d’altres de completament noves. Un aspecte fonamental de la biologia sintètica és el desenvolupament d’eines materials i conceptuals d’ús general (parts biològiques, genomes mínims, cèl·lules artificials, síntesis de DNA…) per a abordar problemes fins ara irresolubles com la biosíntesi de molècules complexes, la descomposició o reciclatge de productes químics tòxics, la detecció biològica d’explosius, la producció biològica d’H2 i altres combustibles, etc. (De Lorenzo et al., 2018). Però aquestes mateixes tècniques permeten també abordar reptes completament innovadors, com la computació amb DNA, el disseny de patrons de desenvolupament, l’ús de bacteris per a eliminar tumors, l’expansió del codi genètic a aminoàcids no naturals i moltes més aplicacions sorprenents (De Lorenzo et al., 2018; O’Day et al., 2019).
«El comportament de components biològics independentment del seu context és un requisit previ per a l’enginyeria de nous dispositius i propietats»
Una premissa recurrent en biologia sintètica és la necessitat d’estandarditzar els components biològics d’una forma separada a què tenen en les seues circumstàncies naturals. El comportament d’aquells components independentment del seu context és un requisit previ per a l’enginyeria de nous dispositius i propietats. Si bé la necessitat i l’oportunitat d’aquesta estandardització han estat identificades clarament, l’èxit de l’esforç ha estat bastant limitat fins ara. Malgrat la llarga llista de parts biològiques depositades en el registre avalat pel MIT, encara hi ha un llarg camí per a complir els requisits que les elevarien al nivell dels estàndards utilitzats per la indústria civil o electrònica (De Lorenzo i Schmidt, 2018). Això és degut en part a la propietat intrínseca de les funcions biològiques d’evolucionar conjuntament com un tot i, per tant, comportar-se d’una manera molt dependent del context. Per tant, existeix la necessitat de desenvolupar millors conceptes i un llenguatge especial per a tractar i classificar aquestes parts biològiques, que es base no sols en la seua possible similitud amb els seus equivalents electrònics, sinó també en una millor comprensió de les funcions biològiques mínimes, principalment relacionades amb la regulació de l’expressió gènica (De Lorenzo i Danchin, 2008). Aquestes millores poden establir les bases d’un futur acord internacional sobre el format d’aquestes parts, la seua disponibilitat i el registre dels seus usuaris.
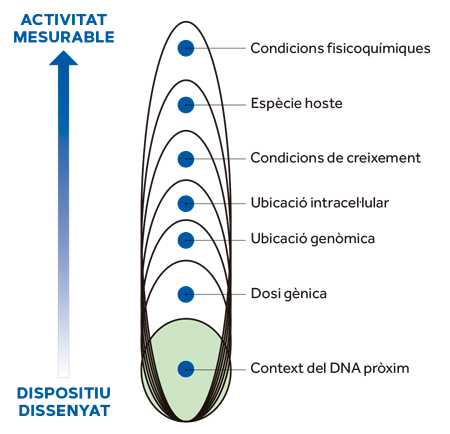
Figura 3. Nivells contextuals de qualsevol construcció genètica. La figura resumeix com la funcionalitat d’una construcció ha de passar per diverses capes de contextos biològics i fisicoquímics abans que l’observador puga mesurar la seua activitat en situacions diferents i comprovar així la seua potencial estandardització. / Font: Porcar, Danchin i De Lorenzo (2014)
Com es mostra en la Figura 3, el funcionament de cada dispositiu d’expressió en una cèl·lula està subjecte almenys a set capes contextuals que van des de la influència mútua immediata de les seqüències de DNA adjacents fins a les condicions fisicoquímiques ambientals. És essencial racionalitzar aquest itinerari des de la composició física fins a la funció final, i d’una manera mesurable i quantificable. Això exigeix tres abordatges convergents. Primer, el modelatge detallat, el mesurament i la parametrització de grans col·leccions de parts funcionals en una varietat de contextos de DNA i condicions de creixement. En segon lloc, la investigació d’un nombre limitat de promotors arquetípics (és a dir, basats en seqüències de DNA consens o molt ben caracteritzats) en diversos entorns genòmics i cel·lulars per a identificar i, en última instància, eliminar els determinants de la dependència del context. I tercer, el disseny avançat de dispositius d’expressió ortogonals (és a dir, independents del context genètic i fisiològic) possiblement basats en parts biològiques reclutades d’elements genètics mòbils (Rao, 2012). El tema dels ribosomes ortogonals i els codis genètics alternatius són àrees d’investigació fascinants que podrien també ser objecte d’un esforç d’estandardització (Wang, Neumann, Peak-Chew i Chin, 2007).
Estàndards α i β per a fer els sistemes vius més fàcils de dissenyar
En el món de l’enginyeria, els termes estàndard i estandardització es refereixen principalment a tres qüestions. En primer lloc, a l’adopció de formes geomètriques específiques i formats de grandària per a l’assemblatge físic dels components d’un sistema fet per l’home. També fan referència a la definició d’unitats de mesura de propietats i paràmetres rellevants, així com les condicions i procediments per a calcular-los (per exemple, amperes per a corrent, ohms per a resistència, etc.). I, per últim, a la implementació de protocols inequívocs per a la fabricació d’objectes. Aquests estàndards permeten l’abstracció de les propietats dels components d’un sistema, la seua descripció precisa amb un llenguatge quantitatiu adequat, també estandarditzat, i la modelització de l’objecte dissenyat amb idèntics mètodes de representació. Un gran avantatge referent a això és la possibilitat de dissociar el disseny detallat d’un producte de la fabricació dels seus components i l’assemblatge final de l’artefacte. Això és comú en l’enginyeria industrial i electrònica, però d’això, quant se’n pot importar a l’àmbit biològic?
«La biologia molecular i la biotecnologia s’han vist afectades des del seu naixement per un menyspreu quasi total pel repte de l’estandardització»
La biologia molecular i la biotecnologia s’han vist afectades des del naixement per un menyspreu quasi total pel repte de l’estandardització. Existeix una nomenclatura generalment caòtica per a gens i eines moleculars. L’absència d’estàndards ha dificultat la mesura comparativa de funcions biològiques tan bàsiques com la força d’un promotor (Beal et al., 2016; Popp, Dotzler, Radeck, Bartels i Mascher, 2017). Per tant, és important examinar quines d’aquestes funcions poden ser objecte d’un esforç d’estandardització que fins i tot podria derivar en una acció prenormativa. Però, què es pot estandarditzar amb el nivell de coneixement que tenim en aquest moment?
Podríem distingir entre estàndards en versió β i α. El primer és el conjunt de regles que adopta una certa comunitat per a millorar la comunicació, la cooperació i la interoperabilitat, però sense pretensions d’universalitat. El punt de partida més comú és l’establiment de cànons per a la composició física de dispositius biològics. Avui dia hi ha múltiples formats que faciliten l’assemblatge de parts de DNA i mòduls individuals per a generar sistemes més complexos (Casini, Storch, Baldwin i Ellis, 2015). Un altre exemple és la introducció de microesferes fluorescents per a calibrar i comparar els experiments de citometria (és a dir, el comptatge i la mitjana de les emissions òptiques de cèl·lules individuals en una població), com s’ha proposat recentment (Beal et al., 2019). Aquests estàndards són necessàriament transitoris, perquè en algun moment l’abaratiment de la síntesi de DNA o l’aparició de noves plataformes de mesura de la fluorescència els farà innecessaris.
«L’adopció de formalismes derivats de l’enginyeria elèctrica i industrial ha estat extraordinàriament útil per al desenvolupament de la biologia sintètica»
Però hi ha un segon tipus d’estàndards (els que he anomenat α) especialment relacionats amb la metrologia de les activitats biològiques que haurien de desenvolupar-se, implementar-se i finalment promulgar-se. Al meu parer, els més importants estan relacionats amb la mesura del flux d’expressió gènica des d’una seqüència codificant de DNA fins a una proteïna. Això falta encara desenvolupar-ho conceptualment i materialment. Les unitats d’RNA polimerasa per segon (PoPS) i de ribosoma per segon (RiPS) com a referència universal per a expressar la força del promotor de la maquinària de traducció són necessàries, però aquestes unitats no s’han abordat seriosament ni des d’un punt de vista fonamental ni com una tecnologia de mesura (Kelly et al., 2009; Sendy, Lee, Busby i Bryant, 2016). Alguns argumenten que la intel·ligència artificial i el disseny assistit per ordinador (CAD) han de ser capaços d’anticipar la majoria dels escenaris possibles dins d’un projecte d’expressió de qualsevol gen d’interès a força de compilar un gran nombre de dades experimentals sobre transcripció, degradació i traducció d’RNA missatgers (Kosuri et al., 2013). Però serà això assolible a curt termini? La traducció és en si mateixa una funció complexa que depèn no sols de la seqüència de l’RNA missatger, sinó també de la seua estabilitat i d’un gran nombre de paràmetres fisiològics com ara la fase de creixement, l’estrès ambiental o la distribució de recursos cel·lulars. La mesura de la velocitat de plegament i les modificacions posttraduccionals són reptes difícilment abordables encara. Potser, com és el cas en altres branques de l’enginyeria, els dissenys biològics del futur dependran per complet de l’aprenentatge automàtic basat en l’experiència (Salis, Mirsky i Voigt, 2009). Però mentrestant, el disseny racional de sistemes vius estarà limitat per l’adopció d’estàndards tant β com α.

Figura 4. Els estàndards necessiten objectes materials de referència. A) Patró del metre. Durant la Revolució francesa, la Convenció Nacional va manar instal·lar mostres de la nova mesura en marbre blanc pels diferents barris de París. En la imatge, patró instal·lat al carrer de Vaugirard. B) Patró del quilo. Aquesta mesura és un altre producte de la Revolució francesa. Com que la definició de quilo com el pes d’un litre d’aigua no era precisa, es va redefinir usant com a referència un objecte cilíndric fabricat amb platí i iridi que es guarda en l’Oficina Internacional de Pesos i Mesures a París. El quilo ha estat redefinit recentment en termes absoluts a través de la mecànica quàntica. En la imatge, rèplica del quilo de l’Institut Nacional de Normes i Tecnologia dels Estats Units, situat a Gaithersburg, a l’estat de Maryland. / Foto esquerra: LPTP / Wikimedia Commons; foto dreta: National Institute of Standards and Technology Digital Collections, Gaithersburg, MD 20899
Què fer?
«Per a descriure, mesurar i “recablar” les funcionalitats biològiques rigorosament, i intercanviar dades, és necessari adoptar estàndards»
És cert que els sistemes biològics no poden equiparar-se automàticament als artefactes humans. Però l’adopció de formalismes derivats de l’enginyeria elèctrica i industrial ha estat extraordinàriament útil per al desenvolupament de la biologia sintètica. La progressió de parts a dispositius i d’aquests a mòduls i sistemes és ara un marc conceptual perfectament acceptat en qualsevol projecte de biologia sintètica. Però per a descriure, mesurar i «recablar» les funcionalitats biològiques rigorosament, i intercanviar dades, és necessari adoptar estàndards (β en el curt termini i α de manera duradora). Més enllà de les regles per a la composició física de les seqüències de DNA, es necessita urgentment el desenvolupament d’un nou tipus de tecnologies que podríem designar com a metrologia biomolecular in vivo. L’objectiu no és només proposar unitats inequívoques per a descriure les corresponents activitats (transcripció, traducció, etc.) sinó també generar entitats materials per a fer-ne el calibratge de referència amb la finalitat de permetre la coordinació de mesuraments en diferents temps i llocs. És interessant que en la història dels estàndards s’haja recorregut a objectes materials (el quilo, el metre, etc.) acuradament guardats com a referència de molts tipus de mesures (Figura 4). Es podrà anar pel mateix camí en el món biològic?
Referències
Andrianantoandro, E., Basu, S., Karig, D. K., & Weiss, R. (2006). Synthetic biology: New engineering rules for an emerging discipline. Molecular Systems Biology, 2(1), 2006.0028. doi: 10.1038/msb4100073
Beal, J., Farny, N. G., Haddock-Angelli, T., Selvarajah, V., Baldwin, G. S., Buckley-Taylor, R., … Workman, C. T. (2019). Robust estimation of bacterial cell count from optical density. BioRxiv, 803239. doi: 10.1101/803239
Beal, J., Haddock-Angelli, T., Gershater, M., De Mora, K., Lizarazo, M., Hollenhorst, J., & Rettberg, R. (2016). Reproducibility of fluorescent expression from engineered biological constructs in E. coli. PLOS ONE, 11(3), e0150182. doi: 10.1371/journal.pone.0150182
Becskei, A., & Serrano, L. (2000). Engineering stability in gene networks by autoregulation. Nature, 405(6786), 590. doi: 10.1038/35014651
Casini, A., Storch, M., Baldwin, G. S., & Ellis, T. (2015). Bricks and blueprints: Methods and standards for DNA assembly. Nature Reviews Molecular Cell Biology, 16(9), 568–576. doi: 10.1038/nrm4014
De Lorenzo, V. (2018). Evolutionary tinkering vs. rational engineering in the times of synthetic biology. Life Sciences, Society and Policy, 14(18). doi: 10.1186/s40504-018-0086-x
De Lorenzo, V., & Danchin, A. (2008). Synthetic biology: Discovering new worlds and new words. EMBO Reports, 9(9), 822–827. doi: 10.1038/embor.2008.159
De Lorenzo, V., & Schmidt, M. (2018). Biological standards for the Knowledge-Based BioEconomy: What is at stake. New Biotechnology, 40, 170–180. doi: 10.1016/j.nbt.2017.05.001
De Lorenzo, V., Prather, K. L., Chen, G. Q., O’Day, E., Von Kameke, C., Oyarzun, D. A., ... Lee, S. Y. (2018). The power of synthetic biology for bioproduction, remediation and pollution control: The UN’s Sustainable Development Goals will inevitably require the application of molecular biology and biotechnology on a global scale. EMBO Reports, 19(4), e4658. doi: 10.15252/embr.201745658
Elowitz, M. B., & Leibler, S. (2000). A synthetic oscillatory network of transcriptional regulators. Nature, 403(6767), 335–338. doi: 10.1038/35002125
Endy, D. (2005). Foundations for engineering biology. Nature, 438(7067), 449–453. doi: 10.1038/nature04342
Galdzicki, M., Rodriguez, C., Chandran, D., Sauro, H. M., & Gennari, J. H. (2011). Standard biological parts knowledgebase. PLoS ONE, 6(2), e17005. doi: 10.1371/journal.pone.0017005
Gardner, T. S., Cantor, C. R., & Collins, J. J. (2000). Construction of a genetic toggle switch in Escherichia coli. Nature, 403(6767), 339–342. doi: 10.1038/35002131
Kelly, J. R., Rubin, A. J., Davis, J. H., Ajo-Franklin, C. M., Cumbers, J., Czar, M. J., ... Endy, D. (2009). Measuring the activity of BioBrick promoters using an in vivo reference standard. Journal of Biological Engineering, 3(1), 4. doi: 10.1186/1754-1611-3-4
Kosuri, S., Goodman, D. B., Cambray, G., Mutalik, V. K., Gao, Y., Arkin, A. P., ... Church, G. M. (2013). Composability of regulatory sequences controlling transcription and translation in Escherichia coli. Proceedings of the National Academy of Sciences, 110(34), 14024–14029. doi: 10.1073/pnas.1301301110
O’Day, E., Hosta-Rigau, L., Oyarzún, D. A., Okano, H., De Lorenzo, V., Von Kameke, C., ... Lee, S. Y. (2019). Are we there yet? How and when specific biotechnologies will improve human health. Biotechnology Journal, 14(1), e1800195. doi: 10.1002/biot.201800195
Popp, P. F., Dotzler, M., Radeck, J., Bartels, J., & Mascher, T. (2017). The Bacillus BioBrick Box 2.0: Expanding the genetic toolbox for the standardized work with Bacillus subtilis. Scientific Reports, 7(1), 15058. doi: 10.1038/s41598-017-15107-z
Porcar, M., Danchin, A., & De Lorenzo, V. (2014). Confidence, tolerance, and allowance in biological engineering: The nuts and bolts of living things. Bioessays, 37(1), 95, doi: 10.1002/bies.201400091
Rao, C. V. (2012). Expanding the synthetic biology toolbox: Engineering orthogonal regulators of gene expression. Current Opinion in Biotechnology, 23(5), 689–694. doi: 10.1016/j.copbio.2011.12.015
Salis, H. M., Mirsky, E. A., & Voigt, C. A. (2009). Automated design of synthetic ribosome binding sites to control protein expression. Nature Biotechnology, 27(10), 946–950. doi: 10.1038/nbt.1568
Sendy, B., Lee, D. J., Busby, S. J., & Bryant, J. A. (2016). RNA polymerase supply and flux through the lac operon in Escherichia coli. Philosophical Transactions of the Royal Society B: Biological Sciences, 371(1707), 20160080. doi: 10.1098/rstb.2016.0080
Wang, K., Neumann, H., Peak-Chew, S. Y., & Chin, J. W. (2007). Evolved orthogonal ribosomes enhance the efficiency of synthetic genetic code expansion. Nature Biotechnology, 25(7), 770–777. doi: 10.1038/nbt1314