Es pot estandarditzar la vida?
Reptes actuals en l’estandardització de la biologia
El concepte d’estàndard ens remet poderosament a la idea de màquines, indústries, dispositius elèctrics o mecànics, vehicles o mobles. De fet, la nostra civilització tecnològica no seria possible –almenys en els termes en què està estructurada avui dia– sense components universals i fiables l’ús generalitzat dels quals permet aconseguir costos competitius i assegurar que els productes siguen més robustos i els seus components, més intercanviables. Per exemple, un caragol de l’Ikea es pot utilitzar en tota una sèrie de mobles estructuralment diferents, i una aplicació es pot executar en molts telèfons intel·ligents diferents. El concepte mateix d’estandardització està vinculat amb la revolució industrial i la producció massiva de béns en cadenes de muntatge. La pregunta que tractarem de respondre en aquest document és fins a quin punt es poden aconseguir estàndards i implementar un procés d’estandardització en l’àmbit biològic.
Paraules clau: dependència del context, modularitat, soroll, promiscuïtat, biologia sintètica.

El concepte d’estandardització s’ha vinculat tradicionalment amb els components universals i intercanviables de dispositius mecànics o electrònics, maquinària, vehicles, mobles, etc. És possible aplicar la finitud, robustesa i universalitat dels estàndards al món de la biologia molecular? / Marco Verch
«El fet que tant els organismes vius com les màquines siguen subjectes a les lleis de la física no significa que els éssers vius siguen màquines»
La biologia sintètica, que podem definir com una aproximació a la biotecnologia des de l’enginyeria, tracta de dotar l’enginyeria genètica d’un caràcter sistemàtic basat en estàndards. La idea central de la biologia sintètica és fer més fàcil el disseny biològic mitjançant la incorporació d’eines i conceptes presos de l’enginyeria (principalment la industrial i l’electrònica). Avui dia, conceptes d’enginyeria com ara abstracció, desacoblament i estandardització s’apliquen al disseny de sistemes biomoleculars. Aquests enfocaments aporten nous coneixements sobre les intricades interaccions moleculars dels sistemes complexos (com les cèl·lules i les interaccions amb el seu entorn), així com avanços en projectes sobre una gran varietat de qüestions, des de l’agricultura fins a la medicina. No obstant això, d’aquest enfocament d’enginyeria per a la biotecnologia sorgeixen molts reptes crítics, la majoria dels quals queden fora de l’abast d’aquest text. Diverses seccions d’aquest article, però, sí que discutiran una d’aquestes qüestions, la que té a veure amb alguns reptes específics que planteja el procés d’estandardització en l’àmbit biològic, amb una mica més de profunditat. El fet que tant els organismes vius com les màquines estiguen sotmesos a les lleis de la física no significa que els éssers vius siguen màquines (per a llegir un treball exhaustiu i recent sobre el tema, vegeu Nicholson, 2019). Les diferències entre els sistemes complexos produïts per l’evolució i els creats per la humanitat estan relacionades amb les dificultats per a estandarditzar els del primer grup, com discutirem en les seccions següents.
Promiscuïtat
Tots els aspectes de la vida són el resultat d’una xarxa d’interaccions entre diversos components, ja siguen molècules intracel·lulars, les cèl·lules dels organismes pluricel·lulars o les espècies d’un ecosistema. Per tant, els éssers vius són sistemes complexos –en el sentit en què s’utilitza el terme en física–. Per exemple, a escala molecular, milers de petites molècules, macromolècules i conjunts supramacromoleculars s’ocupen de construir tots els components cel·lulars a partir de recursos externs (això és, s’ocupen del metabolisme) o de la capacitat d’emmagatzemar i transmetre la informació genètica (digital) que és necessari descodificar per a desenvolupar les funcions cel·lulars. Les proteïnes són les macromolècules clau per a mantenir l’activitat biològica, i els bioquímics i els biòlegs estructurals han reunit una impressionant quantitat de detalls sobre l’arquitectura, dinàmiques i interaccions d’aquests motors moleculars. En el context de la biologia sintètica, les proteïnes se solen considerar parts intercanviables del sistema que, mitjançant interaccions moleculars, poden percebre i processar els senyals i obtenir resultats, com ocorre en qualsevol sistema de processament d’informació. Però els sistemes biològics de disseny presenten diversos problemes que poden provocar una desviació a partir del comportament previst.
«Els sistemes biològics de disseny presenten diversos problemes que poden provocar una desviació a partir del comportament previst»
Quan una proteïna concreta es trasplanta a un context no natiu, pot provocar un gran ventall d’interaccions no desitjades. En els debats sobre les possibles fallades dels sistemes sintètics i reconstruïts, aquesta situació es coneix com errors semàntics (Kittleson, Wu i Anderson, 2012). Per exemple, la sobreproducció d’un metabòlit no natiu, com l’àcid mevalònic, en Escherichia coli provoca efectes tòxics com la inhibició de la biosíntesi d’àcids grassos per part d’un dels intermediaris de l’expressió aliena (Kizer, Pitera, Pfleger i Keasling, 2008). A més de la funció nativa, esculpida per la selecció natural, les proteïnes solen mostrar activitats promíscues; és a dir, reactivitats secundàries no desitjades o interaccions que en condicions normals no representen un mal particular al sistema o són simplement no adaptatives. Les activitats promíscues són responsables del que es coneix com a metabolisme subterrani (D’Ari i Casadesús, 1998), una col·lecció de reaccions secundàries o menors tradicionalment passades per alt pels bioquímics, que són el resultat de l’habilitat dels enzims per a processar substrats diferents als canònics (Khersonsky i Tawfik, 2010). A més, s’han explotat les promiscuïtats moleculars en l’evolució in vitro de noves activitats enzimàtiques, o fins i tot de noves capacitats no naturals, com ara enzims que utilitzen substrats o elements químics no biològics (Arnold, 2019). En la naturalesa, les activitats promíscues també poden representar un inconvenient si generen productes tòxics o inhibidors de funcions essencials. En aquest sentit, cada vegada hi ha més proves de l’existència de molts sistemes de reparació metabòlica –desapercebuts fins fa poc temps– que poden mitigar o evitar aquests problemes de manera eficient (De Crécy-Lagard, Haas i Hanson, 2018).
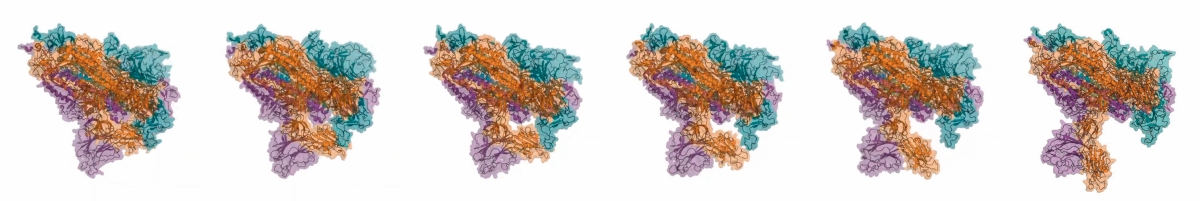
En la biologia sintètica, les proteïnes es consideren parts intercanviables d’un sistema que, mitjançant interaccions moleculars, pot percebre, processar i transmetre informació. No obstant això, poden donar-se interaccions inesperades en els sistemes biològics sintètics, com les activitats promíscues (és a dir, les reaccions i interaccions secundàries, independents de la funció principal de la proteïna) que poden provocar que el sistema funcione de manera incorrecta. Una mostra de la flexibilitat intrínseca de les proteïnes i com posa al descobert funcions la tenim en la proteïna S del coronavirus SARS-CoV-2 (en la imatge): el moviment d’un dels seus dominis estructurals exposa la superfície d’unió amb la proteïna ACE2 humana. Per tot això, un dels objectius de la biologia sintètica ha de ser descobrir les funcions desconegudes de molts enzims codificats en genomes i trobar estratègies per a minimitzar-ne els efectes. / FoldingAtHome
En l’actualitat, el nostre coneixement fragmentari sobre les activitats promíscues representa un greu desavantatge per al disseny de novo dels circuits artificials. La fusió de proteïnes de diferents orígens evolutius en un context no natiu pot provocar l’acumulació d’intermediaris tòxics o no desitjats produïts per les activitats promíscues. En el futur, potser la biologia sintètica responga al repte dels errors semàntics (és a dir, dels efectes secundaris inesperats i indesitjables) amb un esforç conjunt de desenvolupament d’eines per a predir i valorar les activitats promíscues, actualitzant les bases de dades públiques sobre enzims i proteïnes. Tal vegada puga fins i tot crear un mapa complet de la «matèria fosca» metabòlica, és a dir, descobrir la funció desconeguda de molts enzims codificats en els genomes (Ellens et al., 2017), així com dissenyar estratègies per a minimitzar les activitats no desitjades mitjançant el disseny i manipulació de proteïnes i cèl·lules (com, per exemple, dissenyant complexos supramoleculars artificials o compartiments sintètics que canalitzen o emmagatzemen els metabòlits no desitjats). L’objectiu final de totes aquestes línies de treball hauria de ser la descripció detallada d’un comportament estàndard que les biomolècules de disseny han de seguir quan actuen en un context no natiu, una situació que contribuirà a la creació de sistemes artificials més predictius i optimitzats. En qualsevol cas, hem de recordar que les proteïnes són un tipus de matèria tova i que la promiscuïtat és el resultat inevitable de la seua flexibilitat estructural i comportament dinàmic intrínsecs. Per tant, és aconsellable acceptar que la inevitabilitat de la promiscuïtat dels components del sistema és una contingència en qualsevol projecte de biologia sintètica, i avaluar-ne l’impacte sobre el rendiment global del sistema de disseny.
«Les funcions biològiques complexes són el resultat de l’activitat conjunta de poblacions d’entitats, ja siguen molècules, cèl·lules o teixits»
Soroll
A més de la presència de components promiscus, també hem de considerar que les funcions biològiques complexes són el resultat de l’activitat conjunta de poblacions d’entitats, ja siguen molècules, cèl·lules o teixits. En aquest sentit, el comportament estocàstic dels membres individuals de la població s’ha reconegut com una dificultat potencial a l’hora de dissenyar sistemes biològics, i com un inconvenient per a l’estandardització. Fins i tot en poblacions cel·lulars genèticament homogènies, el comportament estocàstic dels individus genera soroll. Aquesta diversitat funcional és deguda a les diferències i fluctuacions en la maquinària molecular disponible dins de cadascuna de les cèl·lules. Fins i tot en condicions ideals, dues cèl·lules poden contenir diferents quantitats de polimerases d’RNA (enzims responsables de copiar cada gen en la plantilla corresponent per a ser descodificat), ribosomes (fàbriques cel·lulars per a la síntesi de proteïnes), factors reguladors (que activen o inhibeixen altres processos) i altres elements. Així, quan les analitzem individualment, les cèl·lules mostren variabilitat funcional (Elowitz, Levine, Siggia i Swain, 2002), i aquest fenomen també s’observa en sistemes dissenyats artificialment. Per exemple, un dels treballs que se sol considerar fundacional de la biologia sintètica, el del disseny, simulació i implementació cel·lular d’un sistema oscil·latori (l’anomenat repressilador, Elowitz i Leibler, 2000), mostrava variabilitat en les cèl·lules individuals a causa de l’estocasticitat dels processos d’expressió gènica.
En la naturalesa, l’efecte dels fenòmens estocàstics i del soroll pot quedar mitigat pel mateix sistema, mentre que a vegades s’utilitzen aquests efectes com a matèria primera per a l’adaptació. En els sistemes biològics de disseny s’han verificat els dos enfocaments (vegeu Kittleson et al., 2012, i les seues referències). De fet, en les últimes dues dècades s’han implementat amb èxit en cèl·lules algunes operacions biocomputacionals estàndard (portes lògiques simples) (Amos i Goñi-Moreno, 2018). No obstant això, queda per respondre la pregunta de si seria possible implementar operacions més complexes dins dels límits de les cèl·lules, amb el seu comportament i soroll intrínsecs. De manera alternativa, diversos grups han fet de la necessitat virtut examinant el desordre biològic i els conjunts multicel·lulars per a comprovar la utilitat d’una àmplia varietat d’enfocaments, com la computació estocàstica, la computació distribuïda i la lògica difusa. L’última frontera seria transcendir el material genètic com a únic suport per als circuits lògics i utilitzar altres nivells d’operació, com les xarxes metabòliques, per a implementar tasques més complexes (per a llegir una discussió exhaustiva sobre els assoliments i reptes de la biocomputació, vegeu Amos i Goñi-Moreno, 2018). En aquest context, seria necessària una definició més relaxada d’estàndard.

El concurs internacional de biologia sintètica iGEM és una gran iniciativa per a educar la pròxima generació de biòlegs sintètics. Sovint, els guanyadors inventen els seus propis models en lloc d’utilitzar els estàndards validats en treballs anteriors. En la imatge, l’espai en què els equips d’estudiants de tot el món mostren els seus projectes als altres durant el concurs. La imatge correspon a l’edició de 2014. / iGEM/Justin Knight
«Els organismes mostren una increïble gamma de variacions, evident en els milions d’espècies que existeixen»
Dependència del context
Existeix un paral·lelisme entre les variants d’un model industrial (un cotxe, per exemple) i les races o llinatges d’animals, plantes o bacteris. Els costos econòmics de canviar la maquinària, les plantilles i els dissenys provoquen un conjunt relativament reduït de variacions dels models de cotxe més populars –o de qualsevol altre artefacte–. Per contra, els organismes mostren una increïble gamma de variacions, evident en els milions d’espècies que existeixen. Per exemple, l’organisme model microbiològic per excel·lència, el bacteri Escherichia coli, inclou milers de soques amb importants diferències genètiques (Moradigaravand et al., 2018). Això ens porta a la pregunta bàsica de si totes les soques es comporten de la mateixa manera des del punt de vista de la biotecnologia. En una investigació duta a terme en un concurs internacional d’estudiants, la competició iGEM, es van transformar sis soques d’E. coli utilitzades habitualment en biologia molecular utilitzant un dispositiu simple consistent en una seqüència genètica promotora i una reportera; és a dir, un circuit genètic simple que produeix un senyal mesurable, normalment en forma de llum fluorescent. Mitjançant el mesurament d’aquests senyals, es va poder identificar l’eficiència del funcionament del circuit en una soca bacteriana en particular. Sorprenentment, es van trobar diferències significatives entre els nivells d’expressió de les sis soques en cinc dels sis dispositius (Vilanova et al., 2015). En un altre experiment del mateix projecte es va transformar una soca d’E. coli utilitzant dos circuits genètics pràcticament idèntics, un que codificava una proteïna roja i un altre que en codificava una de verda. Les cèl·lules amb el fenotip esperat –alhora verd i roig– van resultar ser roges, ja que la major part de la població transformada era roja, verda, o no mostrava fluorescència (Vilanova et al., 2015). Aquests resultats demostren que la gran variabilitat de soques o llinatges en els organismes vius implica una falta de reproductibilitat en el comportament de components genètics teòricament «estàndard» (entesos com a «universals »). La sensibilitat contextual o la influència del context genètic en el rendiment fins i tot del circuit de disseny més simple ha estat reconeguda com un dels grans reptes de la biologia sintètica (Kittleson et al., 2012).
«La gran variabilitat de soques o llinatges en els organismes vius implica una falta de reproductibilitat en el comportament de components genètics teòricament “estàndard”»
El concurs iGEM que hem esmentat anteriorment representa un esforç espectacular per a educar la pròxima generació de biòlegs sintètics. Estudiants de grau de tot el món participen en el concurs i presenten un projecte de biologia sintètica que ha de basar-se, almenys en part, en un sistema de clonatge anomenat «Biobricks™». A causa de l’èxit de les edicions d’iGEM en l’última dècada, Biobricks s’ha començat a usar de manera massiva, per la qual cosa resulten ideals per a realitzar una metanàlisi sobre la reutilització de components en biologia sintètica. L’anàlisi dels equips guanyadors mostra, però, que la majoria tendeixen a desenvolupar els seus propis «estàndards», en lloc d’utilitzar els que ja estan disponibles –validats, en teoria, pels treballs anteriors (Vilanova i Porcar, 2014)–. Això ens porta a la mateixa conclusió irònica expressada prèviament pel premi Nobel Murray Gell-Mann: «Un científic preferiria utilitzar el raspall de dents d’una altra persona abans que la nomenclatura d’un altre científic» (Vilanova i Porcar, 2019).
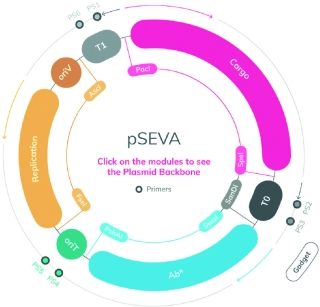
SEVA és un repositori de plàsmids estandarditzats desenvolupat en el Centre Nacional de Biotecnologia. En la imatge, mapa interactiu que mostra l’organització dels vectors SEVA, tal com apareixen en la seua versió web 3.0. Cada plàsmid conté tres mòduls bàsics: una càrrega o porció de DNA (en magenta), un origen de replicació (en groc) i un marcador d’antibiòtic (en blau). Aquests plàsmids poden usar-se per a reconstruir i construir fenotips bacterians complexos. / SEVA/CNB-CSIC
Dit això, hi ha altres exemples de components biològics que s’estan convertint en estàndards de facto en la comunitat de biologia sintètica. La plataforma Standard European Vector Architecture (SEVA) es compon d’una sèrie de plasmidis dissenyats específicament per a ser utilitzats en una varietat d’espècies gramnegatives (i ara també grampositives). Són gratuïts i han estat desenvolupats per l’equip de Víctor de Lorenzo en el Centre Nacional de Biotecnologia (CSIC); s’han enviat un total de 2.019 plasmidis a 35 països i les versions SEVA 2.0 i SEVA 1.0 s’han citat en 88 i 277 ocasions, respectivament (s’acaba de llançar la versió SEVA 3.0, Martínez-García et al., 2020). Això posa en relleu la importància que un component biològic siga robust i útil perquè la comunitat d’usuaris l’adopte com a estàndard.
Modularitat
El concepte de mòdul està connectat amb el d’estàndard. Els dispositius mecànics tenen blocs fàcilment recognoscibles dissenyats per a poder-se muntar, intercanviar o reparar amb facilitat. En biologia, els mòduls són bastant obvis quan ens referim a estructures anatòmiques. Per exemple, no és incorrecte definir els pulmons com a mòduls respiratoris, els músculs com a mòduls motors o de moviment o, en el cas de les cèl·lules eucariotes, considerar els mitocondris com a mòduls interns productors d’energia. De fet, els endosimbionts són un dels millors exemples de mòduls en els sistemes biològics (Porcar, Latorre i Moya, 2013). No obstant això, quan traslladem totes aquestes estructures, ben definides i contingudes en termes físics, a les vies metabòliques (en altres paraules, si deixem el maquinari biològic i ens ocupem del programari), la situació canvia dràsticament, perquè el grau d’intercomunicació, variabilitat, soroll i promiscuïtat enzimàtica present en les vies metabòliques desdibuixa en gran manera el concepte de mòdul. Això té importants conseqüències per a la biologia sintètica, ja que la modularitat implica que un sistema complex es dissenye, construïsca i repare de manera molt més senzilla, perquè podem fraccionar-lo en parts més simples. Si els mòduls no existeixen o estan connectats amb massa complexitat entre ells (això últim és el que ocorre en els sistemes biològics), l’objectiu d’estandarditzar mòduls/blocs/vies es converteix en un gran repte.
«L’estandardització en biologia és un repte immens a causa de la complexitat dels sistemes vius»
Conclusió
En resum, l’estandardització en biologia és un repte immens a causa de la complexitat dels sistemes vius, la seua variació i diversitat intrínseques, la promiscuïtat i soroll de les interaccions i catàlisis de proteïnes, la tendència dels estàndards biològics a dependre del context i l’absència de mòduls biològics fiables. Solucionar aquestes qüestions tècniques és només el primer pas cap a l’estandardització, que és, sobretot, un procés social en el qual els usuaris finals i els responsables polítics consensuen l’elecció i l’ús d’uns estàndards. Com hem vist en aquest treball, encara estem lluny d’aconseguir estàndards en el camp de la biologia, però els èxits parcials (com els plasmidis SEVA) i la magnitud dels beneficis d’estandarditzar la biologia fan que l’esforç valga la pena.
Referències
Amos, M., & Goñi-Moreno, A. (2018). Cellular computing and synthetic biology. En S. Stepney, S. Rasmussen & M. Amos (Eds.), Computational Matter (pp. 93–110). Cham: Springer.
Arnold, F. H. (2019) Innovation by evolution: Bringing new chemistry to life (discurs d’acceptació del Premi Nobel). Angewandte Chemie International Edition, 58(41), 14420–14426. doi: 10.1002/anie.201907729
D’Ari, R., & Casadesús, J. (1998). Underground metabolism. BioEssays, 20(2), 181–186. doi: 10.1002/(SICI)1521-1878(199802)20:2%3C181::AID-BIES10%3E3.0.CO;2-0
De Crécy-Lagard, V., Haas, D., & Hanson, A. D. (2018). Newly-discovered enzymes that function in metabolite damage-control. Current Opinion in Chemical Biology, 47, 101–108. doi: 10.1016/j.cbpa.2018.09.014
Ellens, K. W., Christian, N., Singh, C., Satagopam, V. P., May, P., & Linster, C. L. (2017). Confronting the catalytic dark matter encoded by sequenced genomes. Nucleic Acids Research, 45(20), 11495–11514. doi: 10.1093/nar/gkx937
Elowitz, M. B., & Leibler, S. (2000). A synthetic oscillatory network of transcriptional regulators. Nature, 403(6767), 335–338. doi: 10.1038/35002125
Elowitz, M. B., Levine, A. J., Siggia, E. D., & Swain, P. S. (2002). Stochastic gene expression in a single cell. Science, 297(5584), 1183–1186. doi: 10.1126/science.1070919
Khersonsky, O., & Tawfik, D. S. (2010). Enzyme promiscuity: A mechanistic and evolutionary perspective. Annual Review of Biochemistry, 79, 471–505. doi: 10.1146/annurev-biochem-030409-143718
Kittleson, J. T., Wu, G. C., & Anderson, J. C. (2012). Successes and failures in modular genetic engineering. Current Opinion in Chemical Biology, 16(3-4), 329–336. doi: 10.1016/j.cbpa.2012.06.009
Kizer, L., Pitera, D. J., Pfleger, B. F., & Keasling, J. D. (2008). Application of functional genomics to pathway optimization for increased isoprenoid production. Applied and Environmental Microbiology, 74(10), 3229–3241. doi: 10.1128/AEM.02750-07
Martínez-García, E., Goñi-Moreno, A., Bartley, B., McLaughlin, J., Sánchez-Sampedro, L., Pascual Del Pozo, H., … De Lorenzo, V. (2020). SEVA 3.0: An update of the Standard European Vector Architecture for enabling portability of genetic constructs among diverse bacterial hosts. Nucleic Acids Research, 48(D1), D1164–D1170. doi: 10.1093/nar/gkz1024
Moradigaravand, D., Palm, M., Farewell, A., Mustonen, V., Warringer, J., & Parts, L. (2018). Prediction of antibiotic resistance in Escherichia coli from large-scale pan-genome data. PLOS Computational Biology, 14(12), e1006258. doi: 10.1371/journal.pcbi.1006258
Nicholson, D. J. (2019). Is the cell really a machine? Journal of Theoretical Biology, 477, 108–126. doi: 10.1016/j.jtbi.2019.06.002
Porcar, M., Latorre, A., & Moya, A. (2013). What symbionts teach us about modularity. Frontiers in Bioengineering and Biotechnology, 1, 14. doi: 10.3389/fbioe.2013.00014
Vilanova, C., & Porcar, M. (2014). iGEM 2.0–refoundations for engineering biology. Nature Biotechnology, 32, 420–424. doi: 10.1038/nbt.2899
Vilanova, C., & Porcar, M. (2019). Synthetic microbiology as a source of new enterprises and job creation: A Mediterranean perspective. Microbial Biotechnology, 12, 8–10. doi: 10.1111/1751-7915.13326
Vilanova, C., Tanner, K., Dorado-Morales, P., Villaescusa, P., Chugani, D., Frías, A., ... Porcar, M. (2015). Standards not that standard. Journal of Biological Engineering, 9, 17. doi: 10.1186/s13036-015-0017-9
El treball dels autors ha sigut finançat pel Ministeri de Ciència i Innovació i pels fons del FEDER (RTI2018-095584-B-C41) i de la iniciativa Horitzó 2020 de la Unió Europea (BioRobooST: Fostering synthetic biology standardization through international collaboration, Project ID 210491758).